Implicaciones antropológicas de la presencia del polimorfismo rs9282541 en los warao del delta del Orinoco, Venezuela [1]
Anthropological implications of the presence of the polymorphism rs9282541 in the warao of the Orinoco delta, Venezuela
Implicaciones antropológicas de la presencia del polimorfismo rs9282541 en los warao del delta del Orinoco, Venezuela [1]
Revista Jangwa Pana, vol. 18, núm. 3, pp. 469-481, 2019
Universidad del Magdalena

Esta obra está bajo una Licencia Creative Commons Atribución-NoComercial-CompartirIgual 4.0 Internacional.
Recepción: 31 Enero 2019
Aprobación: 16 Junio 2019
Resumen:
El gen ABCA1 juega un importante papel en la extracción del colesterol intracelular para la formación de la molécula de colesterol HDL. Una variante funcional de este gen, la ABCA1*230Cys (polimorfismo rs9282541), ha sido asociada con diversas alteraciones en los niveles de lípidos en sangre. Se le considera un marcador genético de origen amerindio ya que está presente solamente en nativos americanos y en poblaciones mestizas descendientes de éstos. En ellos se ha observado en frecuencias de hasta un 33 %. Aún no ha sido identificada en poblaciones estudiadas de Europa, Asia o África. Las mayores frecuencias están en Centroamérica, con un decrecimiento hacia los extremos norte y sur del continente. En Venezuela no existen estudios del polimorfismo rs9282541, por lo que no ha sido posible realizar comparaciones con grupos del resto del continente. Con ese interés, el objetivo de la presente investigación fue identificar la frecuencia del ABCA1*230Cys en los warao del delta del río Orinoco (Edo. Delta Amacuro, Venezuela, N=115) y compararla con distancias genéticas reportadas en otros grupos americanos, para aportar información útil a la discusión sobre su origen genético. La frecuencia alélica obtenida fue del 13,05 %, valor intermedio en relación con lo reportado para otros grupos indígenas. Los valores de distancia genética entre warao y el resto de las poblaciones amerindias portadoras del polimorfismo permitieron establecer semejanzas con grupos de origen proto-chibcha y otros cazadores-recolectores del norte del Amazonas de diferente origen lingüístico. La presencia de esa variante amerindia originada en Centroamérica establece una conexión remota con grupos nativos o procedentes de esa región. Estos resultados dan cuenta de la importancia de este tipo de estudios, que integran datos genéticos e históricos, para mejorar el nivel de discusión sobre el origen de las poblaciones indígenas americanas, además de valorar la utilidad de la variante ABCA1*230Cys para establecer vínculos genéticos entre poblaciones de interés. Este es el primer reporte del polimorfismo rs9282541 del gen ABCA1 en poblaciones indígenas venezolanas.
Palabras clave: polimorfismo rs9282541, ABCA1*230Cys, warao, Venezuela.
Abstract:
The ABCA1 gene plays an important role in the extraction of intracellular cholesterol for the formation of the HDL cholesterol. The ABCA1*230Cys allele variant of this gene (polymorphism rs9282541) has been consistently associated with alterations in blood lipid levels. This variant has been observed in maximum frequencies of 33% only in Native Americans and mestizo populations descended from these. This is the reason why it is considered a genetic marker of Amerindian origin. It has not been identified in populations studied in Europe, Asia or Africa. The highest frequencies are shown in Central America, with a decrease toward the extreme north and south of the continent. There are no studies of the rs9282541 polymorphism in Venezuela, therefore it has not been possible to make comparisons with groups from the rest of the continent. With this interest, the objective of this research was to identify the frequency of ABCA1*230Cys in the warao of the Orinoco delta (Delta Amacuro state, Venezuela, N=115), and compare through genetic distances with reports from other American groups. This information is useful to the discussion about its genetic origin. The allele frequency was 13.05%, intermediate value in relation to that reported for other indigenous groups. The genetic distance between warao and the rest of the Amerindian populations establishing similarities with populations of proto-chibchan origin and other hunter-gatherers from the northern Amazon of different linguistic origin. The presence of this Amerindian variant originated in Central America establishes a remote connection with native groups from that region. These results show the importance of such studies integrating genetic and historical data to improve the level of discussion about the origin of Native American populations, also they give value to the usefulness of ABCA1*230Cys variant in other to establish genetic links between populations of interest. This is the first report of the rs9282541 polymorphism of the ABCA1 gene for Venezuelan indigenous populations.
Keywords: polymorphism rs9282541, ABCA1*230Cys, warao, Venezuela.
Introducción
El uso de técnicas genéticas moleculares es cada vez más frecuente para profundizar en el entendimiento de la evolución humana, de los procesos migratorios como el poblamiento de América, y de fenómenos demográficos desde la época prehispánica hasta el presente. En tal sentido, se han descrito numerosas variantes de genes denominadas AIM (Ancestry Informative Markers), que son específicas de algunas poblaciones y tienen la particularidad de marcar el origen étnico/geográfico de algunos grupos humanos (Sampson, Kidd, Kidd y Zhao, 2011; Shriver et al., 2003). Al respecto, el gen ABCA1, cuyo producto o proteína juega un importante papel en la extracción del colesterol intracelular para la formación de la molécula de colesterol HDL (High Density Lipoprotein o lipoproteína de alta densidad), posee polimorfismos o variantes de alelos que han sido asociadas con diversas alteraciones en los niveles de lípidos en sangre (disminución o aumento), que pueden conllevar o predisponer el padecimiento de diabetes, obesidad y enfermedad cardiovascular en individuos de diversas poblaciones humanas alrededor del mundo (Acuña-Alonso et al., 2010; Aguilar-Salinas et al., 2009; Aguilar-Salinas et al., 2011; Dumitrescu et al., 2011; Flores-Dorantes et al., 2010; Hünemeier et al., 2012). Una de ellas, la variante alélica ABCA1*230Cys (c.688C>T), que define el polimorfismo rs9282541, se ha relacionado directamente con niveles bajos de colesterol HDL en sangre, junto a la presencia de sobrepeso y Diabetes Mellitus tipo 2.
Se han publicado datos para 4.634 individuos de diferentes grupos amerindios distribuidos por toda América y para 863 europeos y asiáticos (Acuña-Alonso et al., 2010; Hünemeier et al.,2012). Los reportes indican que la variante ABCA1*230Cys ha sido identificada en la mayoría de los grupos amerindios con una frecuencia promedio de 12 % (alcanzando frecuencias de hasta el 33 %), con valores máximos hacia el centro del continente americano, entre Brasil y México, y un decrecimiento hacia los extremos norte y sur del continente, sin asociación con los grupos lingüísticos. Además, no ha sido identificada en ningún grupo étnico de los otros continentes lo cual indica que, hasta el momento, el ABCA1*230Cys es un alelo exclusivo de poblaciones amerindias o descendientes de ellas y que podría ser empleado como marcador genético tipo AIM (Ancestrality Informative Marker) para la identificación de la ascendencia amerindia de una población.
Se han publicado datos para 4.634 individuos de diferentes grupos amerindios distribuidos por toda América y para 863 europeos y asiáticos (Acuña-Alonso et al., 2010; Hünemeier et al.,2012). Los reportes indican que la variante ABCA1*230Cys ha sido identificada en la mayoría de los grupos amerindios con una frecuencia promedio de 12 % (alcanzando frecuencias de hasta el 33 %), con valores máximos hacia el centro del continente americano, entre Brasil y México, y un decrecimiento hacia los extremos norte y sur del continente, sin asociación con los grupos lingüísticos. Además, no ha sido identificada en ningún grupo étnico de los otros continentes lo cual indica que, hasta el momento, el ABCA1*230Cys es un alelo exclusivo de poblaciones amerindias o descendientes de ellas y que podría ser empleado como marcador genético tipo AIM (Ancestrality Informative Marker) para la identificación de la ascendencia amerindia de una población.
Hünemeier et al. (2012) estimaron la edad de la variante en unos 7540 AP, lo cual es compatible con su origen dentro del continente americano, y proponen una relación gen-cultura. Postulan que dos eventos sucedieron coincidentemente de forma simultánea en el continente americano: el surgimiento de esta variante genética y el del maíz como cultivo (México entre 6300-10000 AP). Los individuos portadores de la variante ABCA1*230Cys se habrían visto favorecidos por una ventaja selectiva, ya que presentan fallas en el transporte reverso del colesterol y, por consiguiente, en la conformación de la molécula de HDL, lo que habría provocado un incremento en los niveles de colesterol intracelular y el almacenamiento este en forma de energía. Esto resultó beneficioso bajo condiciones ambientales de escasez de recursos alimenticios, debido a los cambios estacionales constantes. Actualmente, se observa que tanto las frecuencias más altas como la mayor cantidad de poblaciones portadoras se ubican precisamente en Centroamérica, donde las sociedades son en su mayoría agricultoras y entre sus cultivos principales se destaca el maíz. Este último se convirtió en la fuerza impulsora para el incremento de las frecuencias de ABCA1*230Cys en esta región, para luego dispersarse hacia el sur.
La presencia de esta variante en Suramérica, en algunos casos con frecuencias elevadas en grupos de cazadores-recolectores, es explicado por Hünemeier et al. (2012) como resultado de la existencia de una mayor diversidad en cuanto a hábitats, poblaciones y culturas de Suramérica, en comparación con Mesoamérica. De esa manera, los bajos niveles de flujo génico entre poblados y los reducidos tamaños de población efectiva deben haber favorecido el rol de la deriva génica como fuerza evolutiva impulsora de las frecuencias del polimorfismo rs9282541 en los grupos de esta parte del continente americano, a diferencia de Mesoamérica, donde la selección natural fue la fuerza impulsora de su frecuencia.
En Venezuela no existen estudios del polimorfismo rs9282541, por lo que no es posible realizar comparaciones con grupos del resto del continente americano, que permitan establecer vínculos genéticos. Uno de los grupos indígenas más numerosos del país son los warao; un aspecto a resaltar sobre las investigaciones realizadas en el ámbito de la antropología sobre esta etnia, es la evidente dificultad para establecer un consenso respecto a su origen. Se ha planteado que los warao son descendientes de una población Paleo-Mongoloide que llegó a Suramérica vía el istmo de Panamá y alrededor de 10000 años AP se diferenciaron. Esto sucedió cuando poblaciones de lengua macro-chibcha se dispersaron desde América Central y Colombia hasta el noreste de Venezuela y zonas aledañas, formando sociedades nómadas litorales (Salazar y Vargas, 1992; Schmitz, 1983; Vargas y Sanoja, 1992). Los ancestros de los warao se ubicaron alrededor de 7000 - 6000 AP en los bosques de manglar de los golfos de Paria y Cariaco, y en el valle del río San Juan, en espacios previamente ocupados por caribes y arawak (García-Castro, 2011). Hasta ahora, no hay consenso en relación con la clasificación lingüística warao, pero pudiera tratarse de una filiación lingüística independiente de origen proto-chibcha (Heinen, 2011; Wilbert y Ayala, 2007). En general, los warao forman una cultura de grandes navegantes, pescadores, recolectores y horticultores incipientes, sin cerámica, habitantes de marismas y riberas de caños y ríos. No se han encontrado evidencias arqueológicas directas de los warao por la dificultad que representa su medio ambiente ancestral; carecen de cerámica, debido a lo poco perdurable de su producción material hecha con elementos biodegradables. Por tanto, la ausencia de suficiente evidencia arqueológica hace difícil una aproximación más exacta a la ubicación y cultura warao, previa a la conquista europea (García-Castro, 2011; Wilbert y Ayala, 2007). Debido a las condiciones geográficas, el área central de su hábitat se ha mantenido aislada de otras poblaciones indígenas, así como de la población criolla de Venezuela. Esto favoreció las uniones endogámicas, lo que contribuyó a resguardar su cultura y preservar la singularidad de su lengua, su cosmovisión y sus características genéticas (García-Castro, 2011; Nava, 2012; Wilbert y Ayala, 2007).
Los estudios de marcadores genéticos en warao y poblaciones de lengua chibcha/páez reportados en la literatura internacional comenzaron a finales de la década del 50. Layrisse y Arends (1958) reportaron que los warao del delta del Orinoco en Venezuela presentan frecuencias máximas de 2 % para el alelo Di*a, es decir, para la presencia del antígeno Diego. Estudios posteriores revelaron que estos valores son los más bajos reportados entre las poblaciones indígenas venezolanas (Layrisse y Wilbert, 1999). El factor Diego es un marcador genético de origen asiático que se encuentra distribuido desde Guam y Nueva Guinea en Oceanía, el norte y sureste asiático, hasta América, excepto en la parte baja de Mesoamérica y el norte de Suramérica, donde las frecuencias del factor Diego son muy bajas o iguales a cero entre grupos macro-chibcha, warao y algunos yanomami. A partir de este hallazgo, Layrisse y Wilbert (1999) proponen el origen genéticamente diverso de algunas poblaciones indígenas americanas como los warao, quienes tendrían sus ancestros en grupos asiáticos negativos para el factor Diego (Nava, 2012).
Pocos estudios se han realizado para indagar sobre el origen genético warao, así como la descripción de diferencias y similitudes con otras comunidades indígenas. La mayoría de los realizados son de mediados del siglo XX y evidencian que los warao siguen un patrón genético amerindio, pero arrojan diferencias con otros grupos indígenas venezolanos y americanos.
Estudios con el grupo sanguíneo ABO evidencian que el alelo O está fijado dentro de esta población (Layrisse, Layrisse y Rodríguez-Larralde, 1992), mientras que para el sistema Rhesus (Rh) el haplotipo más frecuente fue el CDe (R1) con más del 60 % (Arends, 1992). En ambos casos se comportan igual que el resto de las poblaciones indígenas suramericanas. Sin embargo, presentan diferencias para otro grupo de polimorfismos de grupos sanguíneos y de proteínas (Arends, 1992; Lugo, 2013).
Estudios más recientes de tipo molecular, como son los marcadores de herencia uniparental para el cromosoma Y, han arrojado una frecuencia de 100 % para el haplogrupo QM3*, característico de poblaciones amerindias y originario del sur de Asia (Bortolini et al., 2003). El ADN mitocondrial también muestra total predominio de haplogrupos indígenas americanos, con 30% de frecuencia para el haplogrupo A2, 58 % para el C1 y del 12 % para el haplogrupo D1 (Izaguirre, Vivenes de Lugo y Castro de Guerra, 2015). Estos resultados tomados en conjunto descartan la posibilidad de cualquier mestizaje significativo no amerindio, elemento que confirma el desarrollo en relativo aislamiento de esta comunidad con respecto a poblaciones europeas o africanas.
En resumen, los resultados disponibles plantean que los warao son una etnia amerindia adaptada a la vida litoral, que avanzó por la Costa Atlántica hasta Venezuela y las Guayanas. Diversas investigaciones han aportado datos que parecieran vincularlos en términos lingüísticos a grupos proto-chibcha (ubicados principalmente en Costa Rica, Panamá y Colombia), mientras que otras presentan datos que por lo general les diferencian, en su acervo genético, de muchas de las poblaciones indígenas de la región, aunque posiblemente vinculadas a poblaciones Diego-negativo de la Baja Mesoamérica (Heinen, 2011; Nava, 2012). Con el interés de obtener más información sobre esta etnia venezolana, el objetivo de este trabajo fue identificar la frecuencia de la variante ABCA1*230Cys en el grupo warao, lo que sumará información útil a la discusión sobre su origen genético.
Materiales y métodos
La población warao
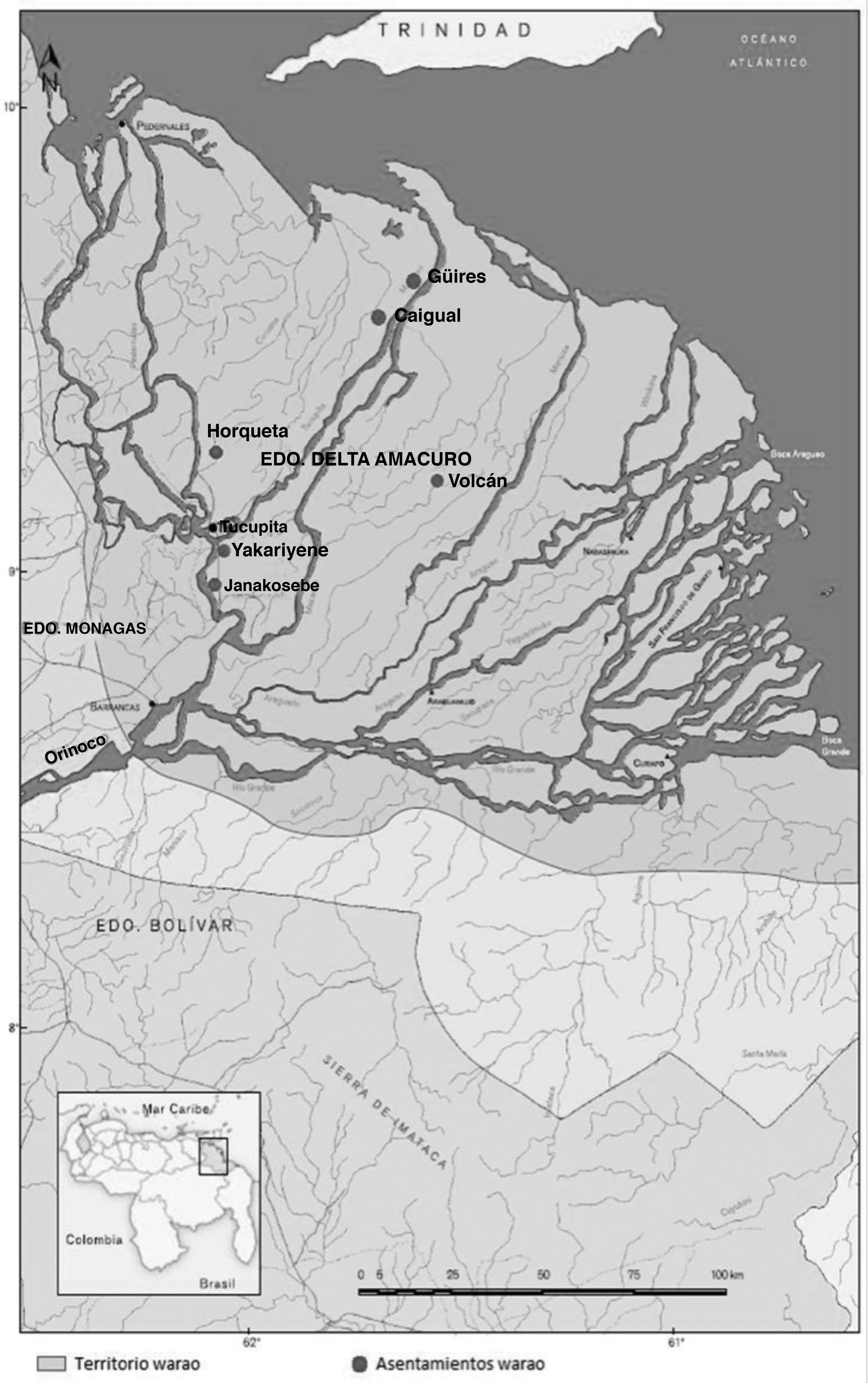
La etnia warao se encuentra distribuida al nororiente de la República Bolivariana de Venezuela, principalmente en el Delta del río Orinoco (Figura 1). También se ubican, aunque en menor proporción, en regiones ribereñas de los estados Monagas, Sucre, Bolívar y en los humedales del noroeste de la Guayana Esequiba (INE, 2011; Nava, 2012; Wilbert y Ayala, 2007). Warao es una autodenominación que significa “gente de canoa” (o gente de tierra baja). Para referirse a los no warao, emplean el término “jotarao”, que significa “gente de tierra alta” (de tierra firme) (Heinen, 2011). El último censo poblacional realizado en el 2011 contabilizó en 48.771 los individuos de esta etnia en Venezuela (INE, 2011), de los que más del 85 % se encuentra asentado en bosques de manglares del delta inferior, en aldeas que alcanzan los 300 habitantes (Nava, 2012).
Muestra
Se estudiaron los ADN de 115 individuos sin parentesco cercano, provenientes de seis comunidades o caseríos de la etnia warao, ubicados en el municipio de Tucupita del estado Delta Amacuro (Figura 1). Los nombres de estas comunidades son Caigual, Güires, Yakariyene, Horqueta, Janakosebe y Volcán. Esta investigación se enmarcó en los proyectos “Estudio de polimorfismos genéticos en individuos del estado Delta Amacuro” y “Estudio de polimorfismos moleculares de interés en genética de poblaciones, salud pública y farmacogenética, en indígenas venezolanos”, los cuales cuentan con la aprobación de la Comisión de Investigación de la UDO (Código CI-2-040102-1306/06), Dirección Regional de Salud, estado Delta Amacuro-Venezuela (oficio 052-2011) y del Comité de Bioética del IVIC (DIR 0575/1512/2014). Los individuos que participaron en el estudio fueron incorporados en los centros rurales de atención médica, donde se contó con la ayuda del personal de salud. Todos los participantes firmaron un consentimiento válido informado. Algunos representantes warao colaboraron con la traducción en los casos en que el participante no hablaba español.
Análisis genético y estadístico
El ADN fue extraído en el laboratorio de Genética Humana del IVIC a partir de sangre completa. Se utilizó el método de extracción salino descrito por Lahiri y Nurnberger (1991), modificado en dicho laboratorio. Para la detección del alelo ancestral ABCA1*230Arg y de la variante de origen amerindio ABCA1*230Cys se utilizó la técnica PCR en tiempo real (qPCR), usando sondas TaqMan, según instrucciones del fabricante (Applied Biosystems).
Las frecuencias alélicas y genotípicas se obtuvieron por contaje directo. Se calculó el equilibrio Hardy-Weinberg con el programa MAXLIK, basado en el método de máxima verosimilitud de Reed y Schull (1968). Se comparó la muestra con otros grupos indígenas americanos a través de distancias genéticas pairwise Fst, usando el programa Arlequin 3.5.1, y esos valores fueron utilizados para elaborar el dendrograma correspondiente mediante el método Neighbor-Joining (Saitou y Nei, 1987) en el editor Drawtree, con el paquete de programas estadísticos PHYLIP 3.6 (Felsenstein, 2004).
Resultados
Se observaron frecuencias genotípicas del 74,79 % para la homocigosis del alelo ancestral ABCA1*230Arg , de 24,34 % para la heterocigosis y 0,87 % para los homocigotos del alelo amerindio ABCA1*230Cys, el cual resultó con una frecuencia del 13,05 % (Tabla 1). El grupo warao se encuentra en equilibrio Hardy-Weinberg para este locus (p≤ 0,05).
A través de distancias genéticas Fst se compararon los warao con otras poblaciones indígenas americanas localizadas en México, Guatemala, Costa Rica, Panamá, Brasil, Paraguay, Colombia, Guayana Francesa, Chile y Bolivia (Acuña-Alonso et al., 2010; Hünemeier et al., 2012).
| Genotipos | n (%) | Alelos | % |
| 230Arg/230Arg | 86 (74,79) | 230Arg | 86,95 |
| 230Arg/230Cys | 28 (24,34) | 230Cys | 13,05 |
| 230Cys/230Cys | 1 (0,87) | - | - |
| Total | 115 (100,0) | Total | 100,0 |
Los valores de distancias obtenidos pueden apreciarse en la Tabla 2, donde también se informa sobre la clasificación lingüística de los grupos aquí considerados (Suárez, 1983; Dixon y Aikhenvald, 2006). Las distancias con diferencias estadísticamente significativas se observaron en relación con los cora, los zapoteca, los maya, los mekranoti, los guaraní y los xavante (p≤ 0,05).
| Población | Distancia Fst con warao | País | Filiación lingüística |
| Suramérica. Sistema económico tradicional: caza/recolección | |||
| Tiriyó , Trio | -0,00030 | Brasil | Caribe |
| Apalaí | -0,01015 | Brasil | Caribe |
| Sateré-Mawé | -0,01183 | Brasil | Tupí |
| Yanomami | -0,01183 | Brasil | Chibcha/independiente |
| Kuben Kran Keng, Kayapó | -0,01642 | Brasil | Je |
| Gorotire , Kayapó | -0,03866 | Brasil | Je |
| Arara | 0,00086 | Brasil | Caribe |
| Parkatejê, Gaviao | 0,00280 | Brasil | Je |
| Wayuu | 0,00709 | Colombia | Arawak |
| Txukahamae , Kayapó | 0,00905 | Brasil | Je |
| Mura, Piraha | 0,01803 | Brasil | Independiente |
| Pacaás-Novos, Wari | 0,02763 | Brasil | Chapacura |
| IçanaRiver , Baniwa | 0,02947 | Brasil | Arawak |
| Xikrin , Kayapó | 0,03151 | Brasil | Je |
| Mekranoti , Kayapó | 0,04803** | Brasil | Je |
| Guaraní | 0,05638** | Brasil | Tupí |
| Palikur | 0,08270 | Guayana Francesa | Arawak |
| Xavante | 0,09862** | Brasil | Je |
| Mesoamérica. Sistema económico tradicional: agricultura | |||
| Tarahumara | -0,00267 | México | Uto -Azteca |
| Totonaco | -0,00438 | México | Totonaco-Tepehua |
| Guaymí | -0,00873 | Costa Rica/Panamá | Chibcha |
| Cabecar | -0,00957 | Costa Rica | Chibcha |
| Mixe | -0,01265 | México | Mixe-Zoque |
| Kaqchiquel -Quiche | -0,01594 | Guatemala | Maya |
| Otomíes | 0,00252 | México | Otomangue |
| Mazahua | 0,00277 | México | Otomangue |
| Nahuatl | 0,00288 | México | Uto -Azteca |
| Teenek | 0,00345 | México | Maya |
| Maya | 0,01520** | México | Maya |
| Yaqui | 0,01717 | México | Uto -Azteca |
| Purepecha | 0,01801 | México | Independiente |
| Zapoteca | 0,02982** | México | Otomangue |
| Cora | 0,06822** | México | Uto -Azteca |
| Andes. Sistema económico tradicional: agricultura | |||
| Huilliche | -0,02083 | Chile | Araucana-Mapuche |
| Ingano | -0,03518 | Colombia | Quechua |
| Aymara | 0,02109 | Chile | Independiente |
| Quechua | 0,02863 | Bolivia | Quechua |
| **Diferencias significativas (p ≤ 0,05) | |||
La Figura 2 corresponde al dendrograma que representa las distancias Fst para los grupos amerindios donde se ha podido identificar la variante ABCA1*230Cys.
Se observan dos agrupamientos: la rama superior, claramente diferenciada del resto, está conformada por poblaciones ubicadas en su mayoría en la región de Centroamérica (México), pero también en Guayana Francesa y Brasil, que es donde se han reportado las frecuencias más altas para el ABCA1*230Cys. La otra ramificación, que incluye el resto de las poblaciones, presenta en su parte inferior a aquellas con las menores frecuencias y, hacia el centro, con valores intermedios, se ubica el grupo estudiado. En general, los warao presentan mayores semejanzas con algunas poblaciones centroamericanas y con brasileñas del norte amazónico.
Según datos reportados, la frecuencia del polimorfismo rs9282541 entre las poblaciones portadoras oscila entre el 2 % y el 33 % (Acuña-Alonso et al., 2010; Hünemeier et al., 2012). Las frecuencias más elevadas se han identificado en Centroamérica, también en la región norte del Amazonas, Guayana Francesa y Brasil. Las frecuencias más bajas se han identificado en poblaciones que residen en la región central de Brasil y más al sur, en la frontera con Argentina y Paraguay. Los resultados de este estudio evidencian que la frecuencia obtenida para los warao se ubica en niveles intermedios (13,05 %). Los amerindios portadores del polimorfismo rs9282541 muestran que las frecuencias más elevadas se han identificado en los cazadores-recolectores palikur de Guayana Francesa (33 %) y xavante del centro de Brasil (31 %), pero también hacia el centro del continente en México, entre los agricultores cora (29 %) y zapoteca 24 %.
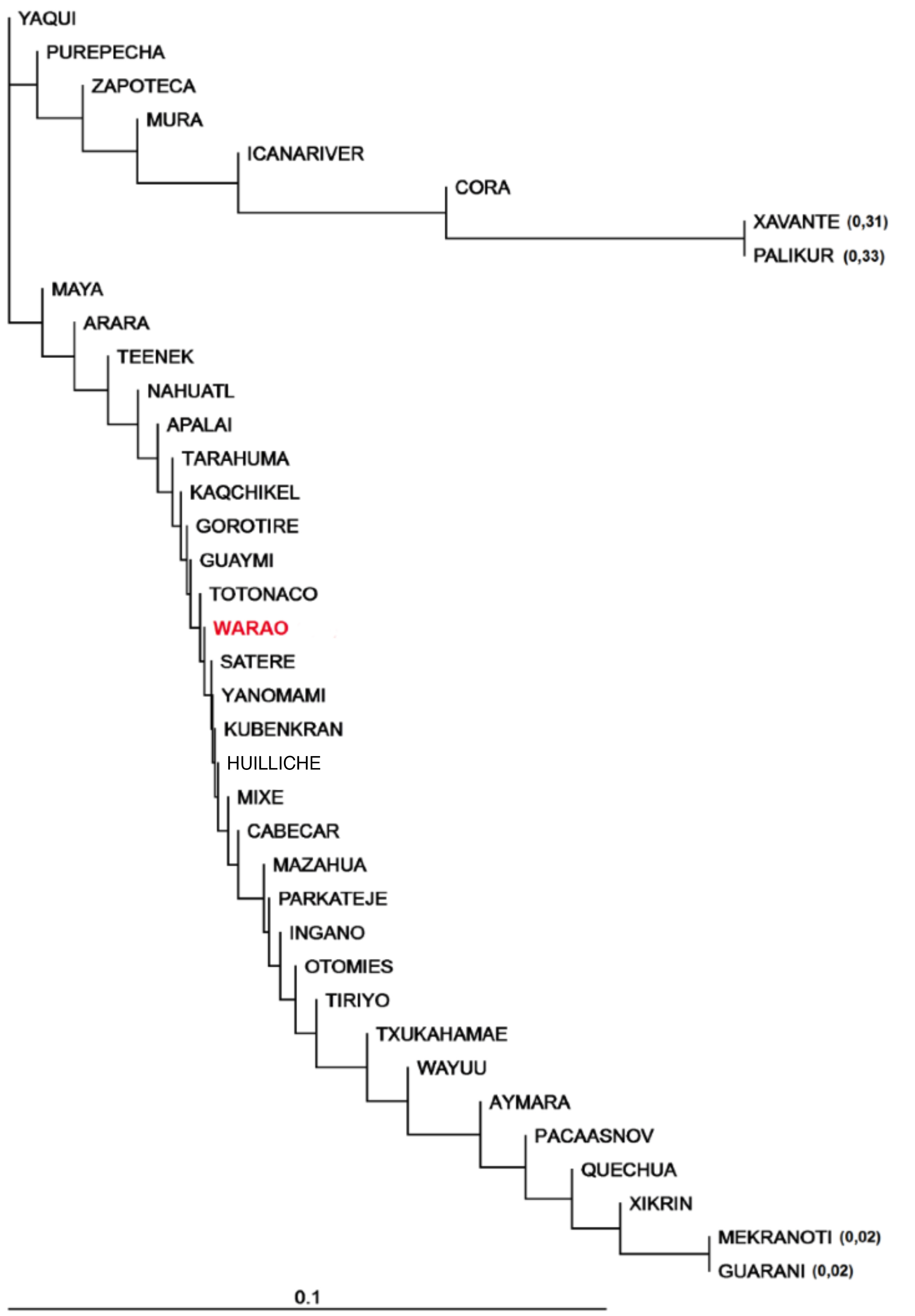
Figura 2
Dendrograma de las 38 poblaciones amerindias en las que se identificó el polimorfismo rs9282541
Elaboración propia
Discusión
El surgimiento de la variante ABCA1*230Cys simultáneamente al proceso de domesticación y consumo del maíz en poblaciones de agricultores de Mesoamérica, representó una ventaja selectiva. Eso explicaría por qué dichos grupos presentan las frecuencias más altas (Hünemeier et al., 2012). Pero esa variante se dispersó hasta Suramérica y está presente en grupos de cazadores-recolectores donde también se reportan frecuencias elevadas, siendo explicadas en esos casos a través de la acción de la deriva génica, debido principalmente por el aislamiento y los pequeños tamaños poblacionales de los grupos de la región. De manera que ese argumento, junto a un efecto fundador, podría explicar la frecuencia observada en los warao, que es un grupo de cazadores-recolectores por tradición.
En una comparación directa, la frecuencia alélica ABCA1*230Cys encontrada en los warao resulta similar a la de los de totonaco (13 %, México), satere-mawé, yanomami, kuben kran keng (12 %, todos en Brasil) y gorotire (14 %, Brasil). Esto se evidencia en el dendrograma que representa la relación genética construida con las distancias entre todos los pares de poblaciones (Fst). En términos geográficos, la mayoría de estos grupos son cercanos a los warao; los yanomami actualmente habitan la frontera entre Venezuela y Brasil, mientras que los grupos brasileños residen al norte del Amazonas. De estos grupos, solo los yanomami podrían compartir un origen lingüístico remoto con los waraos, ya que han sido clasificados también como de lengua aislada o independiente con probable origen proto-chibcha. La distancia genética tan reducida con los cabecar y los guaymí (Costa Rica y Panamá) podría aportar valor a la propuesta de un origen proto-Chibcha para los warao (Acuña-Alonso et al., 2010; Dixon y Aikhenvald, 2006; Hünemeier et al., 2012; Lizot y Kelly, 2007; Nava, 2012; Teixeira, 2005).
Numerosa información histórica establece que el ingreso de grupos humanos al norte de Suramérica ocurrió por el istmo entroamericano de Panamá, a través del cual ciertos grupos proto-chibchas (entre otros) alcanzaron el norte de la península de Paraguaná (Venezuela) y, algunos de ellos, entre los cuales estarían los antepasados warao, se expandieron hasta el extremo oriental del territorio venezolano donde llegaron a compartir espacios con poblaciones arawak y caribes, con quienes tuvieron una convivencia multiétnica y pacífica (Heinen, 2011; Layrisse et al., 1992; Nava, 2012; Salazar y Vargas, 1992; Vargas y Sanoja, 1992; Wilbert y Ayala, 2007).
Estudios genéticos previos con polimorfismos de grupos sanguíneos y proteínas revelan algunas diferencias genéticas de los warao con el resto de los indígenas venezolanos de los que se tiene información (Lugo, 2013). Una diferencia importante se observa con la prevalencia del factor Diego, que alcanza frecuencias de hasta 35 % en indígenas venezolanos, mientras que en este grupo y en los yanomami está prácticamente ausente (Layrisse y Wilbert, 1999; Lugo, 2013; Wilbert y Ayala, 2007). Por otra parte, resultados preliminares de nuestro grupo de investigación muestran diferencias estadísticamente significativas en la distribución de haplogrupos mitocondriales de los warao con las poblaciones indígenas venezolanas de las que se tiene información, siendo no significativas solamente con los yekuana o makiritare (lengua caribe), y los piapoco (lengua arawak), ambos del amazonas venezolano (Izaguirre et al., 2015). De manera que el ADN mitocondrial también sustenta el origen genético diferente de los warao en relación con el resto de los indígenas venezolanos y los posibles intercambios genéticos con los grupos vecinos, de origen caribe y arawak. Bajo el supuesto de un origen único en Mesoamérica para la variante ABCA1*230Cys, su presencia entre los warao establece una conexión remota con grupos originarios o procedentes de esa región, lo que también es sustentado por la inexistencia compartida del alelo Di*a del factor Diego. Mayor número de estudios moleculares se hacen necesarios para mejorar el nivel de discusión sobre el probable origen de esta etnia.
Conclusiones
La frecuencia alélica obtenida fue del 13,05 %, posiblemente producto de la interacción entre efecto fundador, la deriva génica producto de un prolongado aislamiento y la endogamia. Los valores de distancia genética entre warao y el resto de las poblaciones con las cuales se compararon, permitieron establecer semejanzas con grupos chibchas de Panamá, así como con otros del norte del Amazonas, con variado origen lingüístico. Esto no descarta el probable origen proto-chibcha de los warao, ya que la presencia de esa variante amerindia originada en Centroamérica establece una conexión remota con grupos originarios o procedentes de esa región. Estos resultados dan cuenta de la importancia de este tipo de estudios, que integran datos genéticos e históricos, para mejorar el nivel de discusión sobre el origen de las poblaciones indígenas americanas. Además, estos estudios valoran la utilidad de la variante ABCA1*230Cys para establecer conexiones genéticas entre poblaciones de interés y, por otra parte, proporcionan información de utilidad en salud pública. Este es el primer reporte del polimorfismo rs9282541 del gen ABCA1 para poblaciones indígenas venezolanas.
Agradecimientos
Los investigadores agradecen a las personas de la población warao que gentilmente aceptaron participar en esta investigación. A Irene Paradisi, Mary Helen Izaguirre y Luis José Díaz por su apoyo en el trabajo de laboratorio. Los datos presentados constituyen parte de la tesis para obtener el título de antropóloga de Diana Sierra. Financiamiento: proyecto IVIC-899.
Referencias bibliográficas
Acuña-Alonzo, V., Flores-Dorantes, T., Kruit, J., Villarreal-Molina, T., Arellano-Campos, O., Hünemeier, T., Moreno-Estrada, A., … y Cañizales-Quintero, S. (2010). A functional ABCA1 gene variant is associated with low HDL-cholesterol levels and shows evidence of positive selection in Native Americans. Human Molecular Genetics, 19 (14), 2877 - 85.
Aguilar-Salinas, C., Cañizales-Quinteros, S., Rojas-Martínez, R., Roopa, M., Villarreal-Molina, M., Arellano-Campos, O., Riba, L., Gómez-Pérez, F.J., y Tusié-Luna, M. (2009). Hypoalphalipoproteinemia in populations of Native American ancestry: an opportunity to assess the interaction of genes and the environment. Current opinion in lipidology, 20, 92-97.
Aguilar-Salinas, C., Cañizales-Quinteros, S., Rojas-Martínez, R., Roopa, M., Rodriguez-Guillén, R., Ordoñez-Sánchez, M., Riba, L., y Tusié-Luna, M. (2011). The non-synonymous Arg230Cys variant (R230C) of the ATP-binding cassette transporter A1 is associated with low HDL cholesterol concentrations in Mexican adults: A population based nationwide study. Atherosclerosis, 216 : 146-150.
Arends, T. (1992). Estructura genética de la población indígena de Venezuela. Universidad de las Naciones Unidas. Caracas.
Bortolini, M.C., Salzano, F., Thomas, M., Stuart, S., Nasanen, S., Bau, C., Hutz, M., …y Ruiz-Linares, A. (2003). Y-Chromosome Evidence for Differing Ancient Demographic Histories in the Americas. American Journal of Human Genetics, 73 , 524 – 539.
Dixon, R., y Aikhenvald, A. (2006). The amazonian languages . Cambridge Languages Surveys. Cambridge University Press.
Dumitrescu, L., Carty, C.L., Taylor, K., Schumacher, F.R., Hindorff, L.A., Ambite, J., Anderson, G., …y Crawford, D.C. (2011). Genetic Determinants of Lipid Traits in Diverse Populations from the Population Architecture using Genomics and Epidemiology (PAGE) Study. PLoS Genetics 7(6), e1002138. Doi: 10.1371/journal.pgen.1002138
Felsenstein, J. (2004). PHYLIP (Phylogeny Inference Package) version 3.6. Distributed by the author. Department of Genomic Sciences, University of Washington, Seattle, WA.
Flores-Dorantes, T., Arellano-Campos, O., Posadas-Sánchez, R., Villarreal-Molina, M., Medina-Urrutia, A., Romero-Hidalgo, S., Yescas-Gómez, P., …y Cañizales-Quinteros, S. (2010). Association of R230C ABCA1 gene variant with low HDL-c levels and abnormal HDL subclass distribution in Mexican school-aged children. Clinica Chimica, Acta 411 , 1214 - 1217.
García-Castro, A. (2011). Proceso de urbanización de una comunidad warao: El caso La Horqueta Estado Delta Amacuro, Venezuela. (Tesis de doctorado). Instituto Venezolano de Investigaciones Científicas. Caracas, Venezuela.
Heinen, D. (2011). Los warao. En: Lizot, J. (Ed.). Los Aborígenes de Venezuela 3 [pp. 967 – 1113]. Fundación La Salle. Caracas, Venezuela.
Hünemeier, T., Guerra, C., Azevedo, S., Contini, V., Acuña-Alonso, V., Rothhammer, F., Dugoujon, J.M. …y Bortolini, M. (2012). Evolutionary responses to a constructed niche: Ancient mesoamericans as a model of gene-culture coevolution. Revista en línea PLoS ONE; 7(6), e38862. Recuperado de http://www.plosone.org/article/info%3Adoi%2F10.1371%2Fjournal.pone.0038862
Instituto Nacional de Estadística (2011). Primeros resultados de población indígena. Censo Nacional de población y vivienda 2011. Ministerio del poder popular de planificación. Venezuela. Recuperado de http://www.ine.gov.ve/index.php?option=com_content&view=category&id=95&Itemid=9
Izaguirre, M.H., Vívenes de Lugo, M., y Castro de Guerra, D. (2015) Distribución de haplogrupos del ADN mitocondrial en indígenas warao del Estado Delta Amacuro, Venezuela. VI Congreso Científico de la Facultad de Medicina de la LUZ y X Congreso Venezolano de Genética 27 al 30 de abril. Maracaibo, Venezuela. Investigación Clínica 56 (Sup. 1), 832-836. Doi: https://www.redalyc.org/pdf/3729/372940992007.pdf
Lahiri, D.K., y Nurnberger J.L.Jr. (1991). A rapid non-enzymatic method for the preparation of HMW DNA from blood for RFLP studies. Nucleic Acids Research, 19, 5444.
Layrisse, M., y Arends, T. (1958). Estado actual de las investigaciones sobre el Factor Diego. Antropológica. (4), 17-26.
Layrisse, Z., Layrisse, M., y Rodríguez-Larralde, A. (1992). Estudios genéticos en poblaciones de lengua chibcha. Ponencia presentada al VI Congreso de Antropología en Bogotá, Colombia. Junio de 1992. Boletín del Museo del Oro 38-39 .
Layrisse, M., y Wilbert J. (1999). The Diego blood group system and the mongoloid realm. Monograph (44). Caracas: Instituto Caribe de Antropología y Sociología. Fundación la Salle de Ciencias Naturales.
Lizot, J., Kelly, J.A., y Carrera, J. (2007). Los Yanomami. Salud Indígena en Venezuela 1 , 263-379
Lugo, I. (2013). Comparación genética de los grupos Amerindios Barí y Yukpa de la Sierra de Perijá Estado Zulia-Venezuela. (Tesis de pregrado). Caracas: Universidad Central de Venezuela.
Nava, J. (2012). Del morichal a la ribera: dinámicas de cambio cultural y entramados subjetivos entre los warao del delta bajo central. (Tesis de doctorado). Instituto Venezolano de Investigaciones Científicas. Caracas, Venezuela.
Reed, T., y Schull, W. (1968) A general maximum likelihood estimation program. Letters to the editor. American Journal of Human Genetics. 20, 579 - 580.
Saitou, N., y Nei, M. (1987). The Neighbor-joining Method: A new method for reconstructing phylogenetic trees. Molecular Biology Evolution . 4(4), 406 –425.
Salazar, A., y Vargas, A. (1992). Prehistoria de Venezuela: Venezuela antes de la llegada de los europeos. Tropycos. Caracas.
Sampson, J.N., Kidd, K.K., Kidd, J.R., y Zhao, H. (2011). Selecting SNPs to Identify Ancestry. Annals of Human Genetics. 75 (4): 539–553. doi:10.1111/j.1469-1809.2011.00656.x. ISSN 0003-4800. PMC 3141729. PMID 21668909.
Schmitz, P. (1983). Mecanismos das migrações paleoindias na America tropical. 1. O territorio nacional, menos os três Estados sulinos. Cumunidade: XXXV. Reunião Anual da Sociedade Brasileira para o Progresso da Ciência. Brasil.
Shriver, M.D., Parra, E.J., Dios, S., Bonilla, C., Norton, H., Jovel, C., Pfaff, C., Jones, C., Massac, A., Cameron, N., Baron, A., Jackson, T., Argyropoulos, G., Jin, L., Hoggart, C.J., McKeigue, P.M., y Kittles, R.A. (2003). Skin pigmentation, biogeographical ancestry and admixture mapping. Human Genetics 112 (4), 387-99.
Suárez, J. (1983). The mesoamerican indian languages. Cambridge Languages Surveys. Cambridge University Press.
Teixeira, P. (2005). Sateré-Mawé . Retrato de um povo indígena. Universidade Federal do Amazonas. Unicef y UNFPA. Manaus, Brasil. URL https://www.unicef.org/brazil/pt/satere_mawe.pdf
Vargas, I., y Sanoja, M. (1992). La huella asiática en el poblamiento de Venezuela. Lagoven. Caracas.
Wilbert, W., y Ayala, C. (2007). Los warao. Salud Indígena en Venezuela, 2 , 331 - 397.
Notas