CÓDIGOS DE BARRAS GENÉTICOS EN UNA ESPECIE DE Paramacrobiotus (TARDIGRADA: PARACHELA) EN SANTA MARTA, COLOMBIA
GENETIC BARCODING IN A Paramacrobiotus SPECIES (TARDIGRADA: PARACHELA) IN SANTA MARTA, COLOMBIA
Martín Caicedo, Moisés Arquez, Lyda R. Castro y Sigmer Quiroga
Dirección de los autores:
Grupo de Investigación MIKU, Universidad del Magdalena, Carrera 32 No. 22-08, Santa Marta D.T.C.H., Colombia; e-mail: mcamaranto@gmail.com, ORCID: 0000-0001-7802-9573 (M.C.) (S.Q.). Facultad de Ciencias de la Salud, Universidad Simón Bolívar, Sede 2, Carrera 59 No. 59-92, Barranquilla D.E.I.P., Colombia, teléfono: 3444333 Ext. 266; e-mail: marquez2@unisimonbolivar.edu.co, ORCID: 0000-0003-1460-0288 (M.A.). Grupo de Investigación en Evolución, Sistemática y Ecología Molecular (GIESEMOL), Universidad del Magdalena, Carrera 32 No. 22-08, Santa Marta D.T.C.H., Colombia; e-mail: lydaraquelcastro@hotmail. com, ORCID: 0000-0003-3384-3832 (L.R.C.). Programa de Biología, Facultad de Ciencias Básicas, Universidad del Magdalena, Carrera 32 No. 22-08, Santa Marta D.T.C.H., Colombia, teléfono: 4217940 Ext. 3211; e-mail: sigmerquiroga@unimagdalena.edu.co, ORCID: 0000-0002-3321-1360 (S.Q.) (L.R.C.).
DOI: http://dx.doi.org/10.21676/23897864.2125
RESUMEN
Debido a la gran cantidad de especies y a las dificultades en su identificación morfológica, muchos estudios taxonómicos actuales de tardígrados se basan en el uso de técnicas genéticas y moleculares. En Colombia son necesarios estudios enfocados en esta disciplina que permitan complementar la información morfológica; por consiguiente se analizaron siete secuencias del gen COI en el género de tardígrado Paramacrobiotus de la cuenca baja del río Manzanares, en Santa Marta, y se compararon con las secuencias del mismo género presentes en la base de datos de GenBank a través de un análisis de divergencia específica y análisis de vecino más cercano (NJ). Las secuencias obtenidas en el estudio mostraron una clara diferenciación de las secuencias de GenBank y una baja distancia intraespecífica, obteniéndose el código de barras genético correspondiente a una sola especie. Las diferencias de los resultados con el análisis morfológico reflejan la eficacia de técnicas moleculares como los códigos de barras y evidencian la importancia de la integración de técnicas moleculares a los estudios taxonómicos de los tardígrados.
PALABRAS CLAVE: grupo richtersi; umbral de divergencia; citocromo oxidasa I (COI); taxonomía integradora
ABSTRACT
Because of the great number of species and troubles with their morphological identification, many current taxonomic studies in tardigrades are based on the use of genetic and molecular techniques. In Colombia, studies focused in this discipline are needed to be able to complement the morphological information; consequently, seven COI gene sequences were analyzed in the genus of tardigrade Paramacrobiotus at the lower basin of the Manzanares River, in Santa Marta, and were compared with the sequences of the same genus present in the GenBank database through an analysis of specific divergence and neighborjoining (NJ). The sequences obtained in the study showed a clear differentiation of the GenBank sequences, while at the same time showed a low intraspecific distance, obtaining a DNA barcoding for a single species. The differences obtained in comparison with the morphological analyses reflect the effectiveness of molecular techniques such as barcoding, emphasizing the importance of integration of molecular techniques to taxonomic studies on tardigrades.
KEY WORDS: richtersi group; divergence threshold; cytochrome oxidase I (COI); integrative taxonomy
INTRODUCCIÓN
Los tardígrados, conocidos como ositos de agua, son un phylum de organismos microscópicos asociados a microhábitats húmedos, principalmente en la película de agua de musgos y líquenes, aunque también existen especies terrestres, dulceacuícolas y marinas (Bertolani et al., 2004). Actualmente son muy investigados, debido a sus propiedades de criptobiosis, que les permiten sobrevivir a diversas condiciones ambientales extremas (Jönsson, 2007; Schill et al., 2009). Aunque existen más de 1100 especies de tardígrados (Degma et al., 2014), con frecuencia la identificación es difícil, debido a que los caracteres morfológicos no son fáciles de observar y muchos géneros poseen especies con poca variación morfológica (Marley et al., 2011).
En la actualidad, se realizan estudios genéticos y moleculares para la identificación taxonómica basados en métodos como códigos de barras genéticos, unidades taxonómicas operacionales moleculares (MOTU) y secuencias de ARN ribosómico, los cuales han arrojado resultados certeros (Schill y Steinbrück, 2007; Beltrán-Pardo y Bernal-Villegas, 2009). En la última década, los estudios moleculares con tardígrados se han incrementado notablemente, logrando avances en el establecimiento de metodologías y protocolos de trabajo (Schill, 2007; Schill y Steinbrück, 2007; Cesari et al., 2009), así como en la discriminación de nuevos géneros y especies a partir de taxones con características uniformes (Guidetti et al., 2009; Schill et al., 2010).
En Colombia, se han registrado 43 especies (Meyer, 2013; Caicedo, et al., 2014; Lisi et al., 2014). El conocimiento sobre el phylum en el país es incipiente y se requieren avances en la identificación de varias especies aparentemente cosmopolitas. La implementación de técnicas moleculares es una herramienta útil en este tipo de investigaciones, por lo que en el presente trabajo se secuenció y analizó el gen COI en especímenes de tardígrados del género Paramacrobiotus, con el fin de comprobar su eficacia como “código de barra genético” para diferenciar entre especies de este género.
MATERIALES Y MÉTODOS
Se tomaron muestras de musgo en la cuenca baja del río Manzanares (sector de Bonda, Santa Marta, Colombia) entre 46 y 80 m.s.n.m. (11° 13’ 40,2” N - 11° 14’ 02,7” N y 074° 06’ 40,8” W - 074° 06’ 42,7” W). Las muestras fueron guardadas en bolsas de papel para su secado y almacenamiento. Una vez en el laboratorio, las muestras fueron rehidratadas durante 24 horas previas a la revisión, y posteriormente filtradas a través de dos tamices, uno de 1200 μm y otro de 55 μm. Se realizaron dos observaciones en estereoscopio por muestra, entre las 24 y 48 horas posteriores a la rehidratación. Siete especímenes del género Paramacrobiotus fueron aislados para la extracción de ADN y cada uno fue montado previamente en un portaobjetos para su identificación en el microscopio. La información morfológica de los especímenes se encuentra descrita en Caicedo et al. (2014). Cada espécimen fue depositado individualmente en un tubo de 0,5 mL, con 10 μL de agua ultrapura, y almacenado en un congelador a -80 ºC.
Para la extracción, se utilizó el protocolo del kit de purificación de ADN MasterPureTM (Epicentre Biotechnologies). El procedimiento fue realizado de acuerdo con las instrucciones del fabricante, variando solamente los siguientes parámetros:
La incubación a 65 °C se realizó durante dos horas, agitando las muestras cada hora. Posterior a la incubación, a cada muestra se le agregó 1 μL de RNasa A (5 μg / μL) y fueron incubadas a 35 °C durante 30 min. El ADN obtenido al final del procedimiento fue resuspendido en 15 μL de buffer TE.
Se amplificó mediante PCR un fragmento de 640 pb del gen citocromo oxidasa I (COI), utilizando los primers LCO-1490 (Folmer et al., 1994) y HCOoutrev (Schwendinger y Giribet, 2005). Para la amplificación de cada muestra, se diluyeron 2 μL de ADN en 17,5 μL de agua ultrapura y se añadieron 2,7 μL de buffer (10 x), 1,5 μL de MgCl2 (25 mM), 0,2 μL de dNTPs (10 mM), 0,5 μL de cada primer (10 pmol / μL) y 0,1 μL de Taq Polimerasa ABM (5 U / μL). El protocolo de amplificación mediante PCR utilizado fue el siguiente: desnaturalización inicial a 95 ºC por 1 min, 35 ciclos de amplificación (95 ºC por 15 seg, 40 ºC por 1 min y 72 ºC por 1 min 30 seg), elongación final a 72 ºC por 5 min y descenso térmico rápido a 4 ºC. Los productos de PCR fueron verificados con electroforesis en gel agarosa al 2 % y posteriormente purificados con el kit PCR Cleanup Gel Extraction de MACHEREY-NAGEL. El producto resultante fue secuenciado en las dos direcciones por tecnología Sanger.
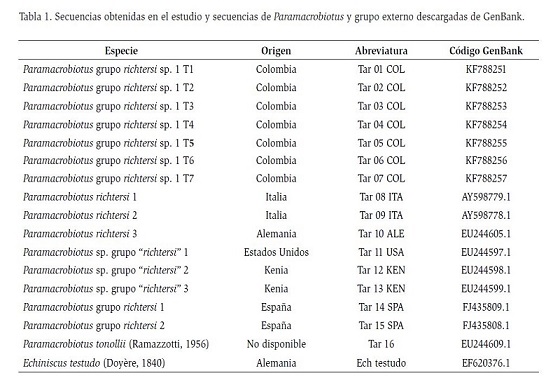
Una vez obtenidas las secuencias, se utilizó el programa ProSeq (Filatov, 2009) para la observación y depuración de los cromatogramas. Las secuencias fueron verificadas a través de la herramienta BLAST del NCBI (Altschul et al., 1990) y posteriormente fueron alineadas con el algoritmo ClustalW, disponible en MEGA V.5 (Tamura et al., 2011). Durante el alineamiento se realizaron correcciones en los marcos de lectura para evitar la presencia de codones de parada que pudieran generar errores en los análisis. Las secuencias obtenidas fueron registradas en GenBank, utilizando la aplicación Sequin, siguiendo las instrucciones del portal web http://www.ncbi.nlm.nih.gov/Sequin/QuickGuide/ sequin.html (NCBI, 2016). Tras su aprobación, se les asignaron los códigos correspondientes (KF788251KF788257) (Tabla 1).

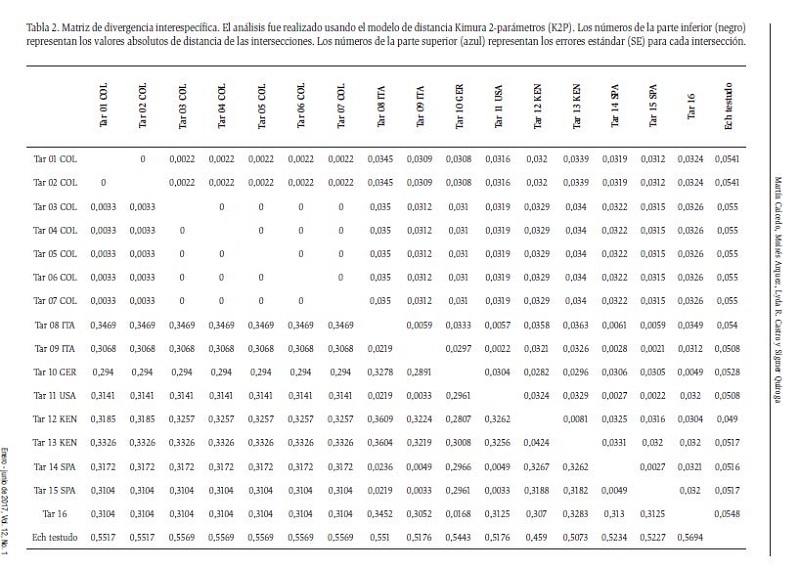
Para calcular las distancias intraespecíficas e interespecíficas de las secuencias se realizó un análisis de divergencia mediante el programa MEGA V.5 (Tamura et al., 2011), utilizando el modelo de distancia de Kimura 2-parámetros, con un bootstrap de 1000 réplicas, registrándose los valores promedio de distancias y los errores estándar (SE). Para diferenciar entre especies con base en los valores de distancia, se tuvo en cuenta un umbral de divergencia del 3 % en el gen COI, propuesto para tardígrados por Cesari et al. (2009). Con dichos valores de distancia, se realizó un análisis de vecino más cercano para obtener la representación gráfica de los patrones de divergencia. En este análisis, se emplearon secuencias de otros ejemplares del mismo género obtenidas de GenBank, para realizar una comparación de las distancias interespecíficas. El heterotardígrado Echiniscus testudo (Doyère, 1840) se escogió como grupo externo (Tabla 1).
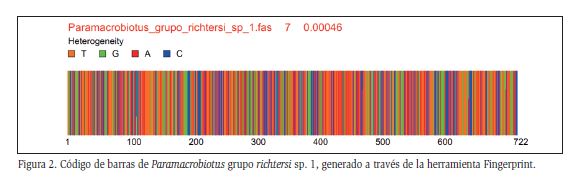
Finalmente, se ilustró la heterogeneidad de sitios específicos dentro del gen COI, mediante una representación de la variación en las secuencias obtenidas a través del programa en línea Fingerprint (Lou y Golding, 2007).
RESULTADOS
En el análisis morfológico inicial, los especímenes recolectados se dividieron en tres posibles especies, teniendo en cuenta diferentes caracteres morfológicos y morfométricos (proporción entre la longitud tubo bucal y el cuerpo, forma de las manchas oculares y proporción de las garras). Después de la corrección de los marcos de lectura y alineamiento, cada una de las secuencias obtenidas tuvo un total de 722 pb, representando una misma especie de tardígrado, Paramacrobiotus grupo richtersi sp. 1, registrada previamente en la región (Caicedo et al., 2014). La composición promedio de nucleótidos fue de T = 36 %, A = 27,5 %, C = 19,5 % y G =17 %.
El análisis de vecino más cercano muestra cuatro grupos que presentan nodos respaldados por valores de bootstrap de 100 % (Figura 1). El primer grupo está representado por las secuencias de P. grupo richtersi sp. 1, sustentando el hecho de que los especímenes pertenecen a la misma especie. El segundo grupo está conformado por dos secuencias, P. richtersi 3 y P. tonollii. El tercer grupo está compuesto por cinco secuencias: las secuencias 1 y 2 de P. richtersi (Italia), la secuencia 1 de P. sp. grupo “richtersi” (Estados Unidos) y las secuencias de P. grupo richtersi (España). Este grupo se etiquetó como secuencias de la región Holártica debido a las ecozonas boreales a las que corresponden. Finalmente, el cuarto grupo congrega a los especímenes de P. sp. grupo “richtersi” 2 y 3 (Kenia).

El análisis de distancias de las secuencias obtenidas mostró distancias intraespecíficas muy bajas, entre 0 % (Tar 01 COL-Tar 02 COL) y 0,33 % (Tar 01 COL-Tar 03 COL a Tar 07 COL). El promedio de las distancias entre las secuencias fue de 0,14 %. Comparando dichos valores con el umbral de divergencia del gen COI en tardígrados (Cesari et al., 2009), puede afirmarse que las siete secuencias pertenecen, en efecto, a una misma especie. Las variaciones de las distancias interespecíficas presentaron un margen amplio, desde 4,24 % (Tar 13 KEN-Tar 12 KEN) hasta 56,94 % (Ech testudo-Tar 16) (Tabla 2). El promedio de estas distancias fue de 26,78 %. Entre los macrobiótidos, la mayor distancia fue de 36,09 % (Tar 12 KEN-Tar 08 ITA).
El código de barras obtenido mediante Fingerprint, ilustra de forma gráfica la composición de nucleótidos de la especie analizada que evidencia un alto porcentaje de timina y adenina en las secuencias, además de uniformidad de las secuencias entre los siete especímenes. El valor de la longitud de las barras en las secuencias fue de 0,00046, cifra que refleja la similitud de las secuencias a un nivel intraespecífico (Figura 2).
DISCUSIÓN
A partir de un estudio morfológico y molecular del eutardígrado Macrobiotus macrocalix Bertolani y Rebecchi (1993), se propuso un umbral de divergencia del 3 % para distinguir entre las especies de tardígrados, un valor semejante al de otros grupos de invertebrados (Cesari et al., 2009). Las secuencias generadas en el presente estudio mostraron valores de divergencia muy bajos (0 - 0,42 %), ajustándose dentro del intervalo de distancias intraespecíficas. Dichos resultados fueron similares a los valores obtenidos en otras investigaciones con códigos de barras generados a partir del COI en tardígrados (Cesari et al., 2009, 2013; Bertolani et al., 2011).
Inicialmente, las secuencias fueron subdivididas en tres posibles especies diferentes, teniendo en cuenta algunas características morfológicas y morfométricas; sin embargo, durante el proceso de montaje en placas, los tardígrados pueden sufrir ocasionalmente alteraciones debido al proceso de fijación, por lo cual se debe tener cuidado al seleccionar rasgos morfológicos para identificación (O. Lisi, com. pers.). Los resultados observados en el presente estudio confirman la utilidad e importancia de complementar la identificación morfológica con análisis moleculares.
El análisis de las secuencias obtenidas mostró resultados contrastantes con las secuencias descargadas de GenBank. Cada una de las secuencias del segundo grupo (P. tonollii y P. richtersi 3) pertenece a un subgrupo de especies de Paramacrobiotus con claras diferencias morfológicas como los placoides faríngeos y la morfología de los huevos (Pilato y Binda, 2010). Sin embargo, los análisis moleculares mostraron que corresponden a la misma especie. Tales resultados indican que pudo ocurrir un error en la identificación morfológica de los especímenes utilizados en la secuenciación. Es necesario realizar una revisión rigurosa en la identificación de dichos especímenes y complementar con otros análisis filogenéticos, a fin de obtener conclusiones sólidas.
Cinco secuencias de tres países (España, Estados Unidos e Italia) presentaron valores de divergencia por debajo del umbral propuesto (Cesari et al., 2009), sugiriendo que pertenecen a una misma especie. Si se confirma que se trata de P. richtersi, y se realizan otros estudios de taxonomía integradora en otras regiones, es posible que pueda redefinirse la distribución de esta especie como holártica, lo cual resolvería definitivamente su estatus actualmente dudoso como especie cosmopolita (Michalczyk et al., 2012). En contraste, si bien las secuencias de Kenia posiblemente están relacionadas con Paramacrobiotus kenianus (Schill et al., 2010), los diferentes valores de divergencia indican que puede tratarse de dos especies diferentes. No obstante, dado que P. kenianus es una especie descrita casi exclusivamente a partir de datos moleculares del espaciador interno terminal 2 (ITS2), se requieren estudios taxonómicos que utilicen varios marcadores y caracteres morfológicos importantes.
El gen COI es una fuente de información muy útil para la diferenciación entre especies muy cercanas de tardígrados, o grupos de especies con escasas diferencias morfológicas (Cesari et al., 2009; Bertolani et al., 2011). Con el fin de generar relaciones filogenéticas robustas, es necesario integrar su información con otros genes (18S, 28S e ITS2) (DeSalle et al., 2005) y con la información morfológica (Guidetti et al., 2009; Bertolani et al., 2011; Guil y Giribet, 2012). Se recomienda un estudio de taxonomía integradora, usando tanto técnicas de caracterización morfológica como análisis moleculares, que permita tener unas bases claras para la diferenciación correcta de las especies seleccionadas, particularmente en géneros con especies crípticas o grupos de especies, aplicando técnicas como preparación de vouchers de especímenes hologenóforos (Bertolani et al., 2011) y cultivo de especies (Kagoshima et al., 2013).
CONCLUSIONES
El estudio mostró que un código de barras genético basado en genes como el COI es una buena herramienta para una correcta identificación de especies de tardígrados, especialmente ante la presencia de dificultades en la identificación morfológica. Los futuros estudios taxonómicos de tardígrados deben combinar el uso de caracteres morfológicos y técnicas moleculares, considerando diversos genes, a fin de obtener un respaldo robusto para los resultados.
AGRADECIMIENTOS
Los autores desean extender sus agradecimientos a los grupos de investigación MIKU y GIESEMOL, de la Universidad del Magdalena, por su ayuda, apoyo y recomendaciones durante las fases de muestreo y análisis de especímenes y datos moleculares. Igualmente, agradecimientos a los doctores Oscar Lisi (Universidad de Catania-Sicilia) y Noemí Guil (Museo Nacional de Ciencias Naturales-España) por sus sugerencias y recomendaciones durante la realización del estudio.
BIBLIOGRAFÍA
Altschul, S.F., Gish, W., Miller, W., Myers, E.W. y Lipman, D.J. 1990. Basic local alignment search tool. Journal of Molecular Biology 215: 403–410.
Beltrán-Pardo, E. y Bernal-Villegas, J. 2009. Acercamiento a los estudios actuales sobre el filo Tardigrada y su importancia en la medicina. Universidad de Medicina de Bogotá 50: 380–403.
Bertolani, R. y Rebecchi, L. 1993. A revision of the Macrobiotus hufelandi group (Tardigrada, Macrobiotidae), with some observations on the taxonomic characters of eutardigrades. Zoologica Scripta 22(2): 127–152.
Bertolani, R., Guidetti, R., Jönsson, K.I., Altiero, T., Boschini, D. y Rebecchi L. 2004. Experiences with dormancy in tardigrades. Journal of Limnology 63: 16–25.
Bertolani, R., Rebecchi, L., Giovannini, I. y Cesari, M. 2011. DNA barcoding and integrative taxonomy of Macrobiotus hufelandi C.A.S. Schultze 1834, the first tardigrade species to be described, and some related species. Zootaxa 2997: 19–36.
Caicedo, M., Londoño, R. y Quiroga, S. 2014. Catálogo taxonómico de los ositos de agua (Tardigrada) de la cuenca baja de los ríos Manzanares y Gaira, Santa Marta, Colombia. Boletín Científico Museo de Historia Natural Universidad de Caldas 18(1): 197–209.
Cesari, M., Bertolani, R., Rebecchi, L. y Guidetti, R. 2009. DNA barcoding in Tardigrada: the first case study on Macrobiotus macrocalix Bertolani y Rebecchi 1993 (Eutardigrada, Macrobiotidae). Molecular Ecology Resources 9: 699–706.
Cesari, M., Bertolani, R., Rebecchi, L., Giovannini, I. y Guidetti, R. 2013. A DNA barcoding approach in the study of tardigrades. Journal of Limnology 72(1): 182–198.
Degma, P., Bertolani, R. y Guidetti, R. 2014. Actual checklist of Tardigrada species (2009-2014, Ver. 27: 31-102014)._URL:_http://www.tardigrada.modena.unimo.it/ miscellanea/Actual%20checklist%20of%20Tardigrada. pdf. Consultado: 4 de diciembre de 2014.
DeSalle, R., Egan, M.G. y Siddall, M. 2005. The unholy trilogy: taxonomy, species delimitation and DNA barcoding. Philosophical Transactions of the Royal Society 360: 1905–1916.
Doyère, L.M.F. 1840. Memoire sur les Tardigrades. I. Annales des Sciences Naturelles, Paris 2(14): 269–362.
Filatov, D.A. 2009. Processing and population genetic analysis of multigenic datasets with ProSeq3 software. Bioinformatics 25: 3189–3190.
Folmer, O., Black, M., Hoeh, W., Lutz, R. y Vrijenhoek, R. 1994. DNA primers for amplification of mitochondrial cytochrome C oxidase subunit I from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology 3: 294–299.
Guidetti, R., Schill, R.O., Bertolani, R. y Dandekar, T., Wolf, M. 2009. New molecular data for tardigrade phylogeny, with the erection of Paramacrobiotus gen. nov. Journal of Zoological Systematics and Evolutionary Research 47: 315–321.
Guil, N. y Giribet, G. 2012. A comprehensive molecular phylogeny of tardigrades-adding genes and taxa to a poorly resolved phylum-level phylogeny. Cladistics 28: 21–49.
Jönsson, K.I. 2007. Tardigrades as a Potential Model Organism in Space Research. Astrobiology 7(5): 757–767.
Kagoshima, H., Imura, S. y Suzuki, A.C. 2013. Molecular and morphological analysis of an Antarctic tardigrade, Acutuncus antarcticus. Journal of Limnology 72(1): 15–23.
Lisi, O., Londoño, R. y Quiroga, S. 2014. Tardigrada from a sub-Andean forest in the Sierra Nevada de Santa Marta (Colombia) with the description of Itaquascon pilatoi sp. nov. Zootaxa 3841(4): 551–562.
Lou, M. y Golding, G.B. 2007. FINGERPRINT: visual depiction of variation in multiple sequence alignments. Molecular Ecology Resources 7(6): 908–914.
Marley, N.J., McInnes, S. y Sands, C.J. 2011. Phylum Tardigrada: A re-evaluation of the Parachela. Zootaxa 2819: 51–64.
Meyer, H. 2013. Terrestrial and freshwater Tardigrada of the Americas. Zootaxa 3747(1): 001–071.
Michalczyk, Ł., Welnicz, W., Frohme, M. y Kaczmarek, Ł. 2012. Redescriptions of three Milnesium Doyère, 1840 taxa (Tardigrada: Eutardigrada: Milnesiidae), including the nominal species for the genus. Zootaxa 3154: 1–20.
NCBI Resource Coordinators. 2016. Database resources of the National Center for Biotechnology Information. Nucleid Acids Research 44(D1): D7–D19.
Pilato, G. y Binda, M.G. 2010. Definition of families, subfamilies, genera and subgenera of the Eutardigrada, and keys to their identification. Zootaxa 2404: 1–54.
Schill, R.O. 2007. Comparison of different protocols for DNA preparation and PCR amplification of mitochondrial genes of tardigrades. Journal of Limnology 66(1): 164–170.
Schill, R.O. y Steinbrück, G. 2007. Identification and differentiation of Heterotardigrada and Eutardigrada species by riboprinting. Journal of Zoological Systematics and Evolutionary Research 45(3): 184–190.
Schill, R.O., Mali, B., Dandekar, T., Schnölzer, M., Reuter, D. y Frohme, M. 2009. Molecular mechanisms of tolerance in tardigrades: New perspectives for preservation and stabilization of biological material. Biotechnology Advances 27: 348–352.
Schill, R.O., Förster, F., Dandekar, T. y Wolf, M. 2010. Using compensatory base change analysis of internal transcribed spacer 2 secondary structures to identify three new species in Paramacrobiotus (Tardigrada). Organisms Diversity and Evolution 10(4): 287–296.
Schwendinger, P.J. y Giribet, G. 2005. The systematics of the south-east Asian genus Fangensis Rambla (Opiliones: Cyphophthalmi: Stylocellidae). Invertebrates Systematics 19: 297–323.
Tamura, K., Peterson, D., Peterson, N., Stecher, G., Nei, M. y Kumar, S. 2011. MEGA5: Molecular Evolutionary Genetics Analysis using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods. Molecular Biology and Evolution 28: 2731–2739.
Fecha de Recibido: 18/02/2016
Fecha de Aceptación: 05/06/2017
Fecha de Aceptación: 22/06/2017
Para citar este artículo: Caicedo, M., Arquez, M., Castro, L.R. y Quiroga, S. 2017. Códigos de Barras Genéticos en una especie de Paramacrobiotus (Tardigrada: Parachela) en Santa Marta, Colombia. Revista Intropica Vol. 12(1): 15 -22. DOI: http://dx.doi.org/10.21676/23897864.2125