ESTANDARIZACIÓN DEL ANÁLISIS MULTI-LOCUS DE NÚMERO VARIABLE DE REPETICIONES EN TÁNDEM PARA EL ESTUDIO DE Staphylococcus aureus RESISTENTES A METICILINA AISLADOS DE LA COMUNIDAD EN PARAGUAY
MULTIPLE-LOCUS VARIABLE-NUMBER OF TANDEM REPEAT ANALYSIS STANDARDIZATION FOR COMMUNITY ISOLATES METHICILINERESISTANT Staphylococcus aureus STUDY IN PARAGUAY
TÍTULO CORTO: ESTANDARIZACIÓN DEL ANÁLISIS MULTI-LOCUS DE NÚMERO VARIABLE
RESUMEN
Staphylococcus aureus es un patógeno capaz de causar infecciones con amplio rango de severidad y adaptarse a diferentes tejidos. Su epidemiología es compleja, por circulación de cientos de clones a nivel mundial, lo que requiere de métodos moleculares reproducibles y de alto poder discriminatorio para la identificación de los mismos. El presente estudio tuvo como objetivo principal la estandarización del análisis multi-locus de número variable de repeticiones en tándem (MLVA) para análisis de variabilidad genética de aislados de S. aureus previamente tipificados por electroforesis en gel de campo pulsado (PFGE), gold standard para tipificación de aislados. La MLVA se realizó por amplificación de 7 locus VNTR (clfA, clfB, sdrC, sdrD, sdrE, sspA y spA) por PCR. Se alcanzó un alto nivel de reproducibilidad. El empleo de cepas previamente tipificadas por análisis de secuencias multi-locus (MLST), PFGE, locus spa y cassette SCCmec, permitió validar de forma comparativa el agrupamiento generado por MLVA. Los aislados que fueron agrupados como idénticos por MLVA presentaron resultados congruentes con la totalidad de las otras técnicas moleculares y esta demostró ser más sensible que PFGE para distinguir entre aislados que presentaron patrones PFGE idénticos. La MLVA cumple todos los criterios de un método de tipificación útil.
Palabras Clave: Staphylococcus aureus; epidemiología; técnicas de tipificación bacteriana; tipificación molecular.
ABSTRACT
Staphylococcus aureus is a pathogen that can produce several infections with a wide range of severity and it has the ability to adapt to different tissues. The epidemiology is complex, due to circulation of many different clones worldwide, so the analysis for its identification requires reproducible and high discriminatory power molecular methods. The aim of this study was to standardize the molecular technique multiple-locus variable number of tandem repeat analysis (MLVA) for the genetic variability analysis of S. aureus isolates, previously characterized by pulsed field gel electrophoresis (PFGE). The MLVA was made by PCR amplification of seven VNTR locus (clfA, clfB, sdrC, sdrD, sdrE, sspA y spA). A high level of reproducibility has been reached in the study. The use of isolates previously typified by multi-locus sequence typing (MLST), PFGE, locus spa and cassette SCCmec, allowed to validate the MLVA clusters comparatively. The isolates that were clustered by MLVA as the same isolate, showed the same results by other molecular techniques, and the MLVA can distinguish isolates with identical PFGE patterns. This technique meets all the criteria of a useful molecular typification technique.
Keywords: Staphylococcus aureus; epidemiology; bacterial typing techniques; molecular typing.
INTRODUCCIÓN
Staphylococcus aureus es un microorganismo capaz de causar infecciones con un amplio rango de severidad debido a su alta capacidad patogénica, su habilidad para colonizar exitosamente, adaptarse y sobrevivir en diferentes tejidos celulares durante la infección1-3.
Los métodos de tipificación molecular permiten la comprensión de la variabilidad genética de los microorganismos, e incluyen a la electroforesis en gel de campo pulsado (PFGE) y los basados en la reacción en cadena de la polimerasa (PCR), dirigidos a la amplificación de elementos repetitivos. La PFGE es un método de alto poder discriminatorio por basarse en el análisis del polimorfismo de fragmentos genómicos; pero tiene la desventaja de ser un método laborioso y de requerir mucho tiempo para su realización, así como equipamientos específicos y costosos4,5.
La metodología MLVA (Multiple-Locus Variable-Number Tandem Repeat Analysis) discrimina entre diferentes aislados clínicos de S. aureus basada en el análisis de siete loci individuales (clfA, clfB, sdrC, sdrD, sdrE, sspA y spA) denominados VNTRs (Variable-Number Tandem Repeat). La amplificación de estos loci mediante una PCR múltiple genera un patrón de bandas característico para cada aislado, permitiendo establecer un relacionamiento evolutivo entre aislados, con alto poder discriminatorio y buenos niveles de reproducibilidad interlaboratorial comparable a la PFGE4,6-8.
En Paraguay la PFGE es poco accesible debido al alto costo, por lo que la información existente sobre la diversidad genética de aislados de S. aureus en el país es muy escasa9. Weiler10 describió, en el 2011, un brote epidémico asociado al consumo de leche ultrapasteurizada contaminada con S. Aureus, el mismo afectó a 400 personas de diferentes regiones del país, incluso se registró un óbito de un lactante en este caso; los aislados fueron analizados por PFGE en el exterior, identificando a la leche como fuente de intoxicación y a un operario de la línea de producción como origen de la contaminación. Este fue el primer brote de ETA denunciado en nuestro país (Paraguay), en el cual se pudo aislar, caracterizar y tipificar molecularmente en el extranjero, con una demora de un par de años, el agente etiológico10.
El presente estudio tuvo como objetivo principal la estandarización de la técnica molecular MLVA para análisis de variabilidad genética de aislados de S. aureus previamente tipificados por el gold standart (PFGE). La puesta a punto de la técnica MLVA permitiría un screening de gran cantidad de aislados en caso de sospecha de brotes, con lo que se generaría una respuesta a tiempo real que permita tomar medidas apropiadas en estos casos.
MATERIALES Y MÉTODOS
Diseño del estudio y población
Prueba diagnóstica, con muestreo no probabilístico de conveniencia. Se emplearon 11 aislados de Staphylococcus aureus resistentes a meticilina aislados de la comunidad (SARM-CO), caracterizados previamente tanto fenotípica como genotípicamente, que se encontraban criopreservados en el biobanco de muestras del IICSUNA. La fenotipificación consistió en el antibiograma por métodos de difusión Kirby-Bauer, empleando discos de la marca Oxoid/BBI siguiendo los criterios del CLSI (Clinical and Laboratory Standards Institute). Todos los aislados empleados en este estudio eran resistentes a meticilina, ensayados por métodos de difusión en disco de cefoxitina y sensibles a vancomicina, ensayados por el método E-test (por detección de la concentración inhibitoria mínima). La genotipificación previa incluyó la detección de los genes mecA, luk-PV, sea, seb, sec, sed, seh, hla, hlb, eta y etb11.
El Spa typing se llevó a cabo empleando oligonucleótidos descritos por Shopsin et al12 y la posterior secuenciación en Macrogen Inc. (Corea). Para la identificación de los tipos spA se recurrió al Spa DataBase (www.spaserver. ridom.de). La detección del cassette SCCmec se efectuó según lo descrito por Kondo et al13. El análisis MLST se hizo en un aislado meticilino resistente representativo de cada pulsotipo, empleando oligonucleótidos descritos por Enright et al14. Las secuencias obtenidas se compararon con la base de datos de MLST.
La PFGE se realizó siguiendo el protocolo de Chung et al15. En el proceso se incluyeron clones conocidos (clon CAA, pulsotipo A-ST5-t311-IV-PVL+ y el clon pulsotipo C- ST30t019-IV-PVL+). Se construyó un dendograma empleando el software Treecon 1.3b, aplicando el algoritmo de cluster (UPGMA, applying unweighted pair-group method clustering analysis) y el coeficiente de Dice. Aislados con patrones de bandas similares >85% fueron considerados pertenecientes al mismo pulsotipo.
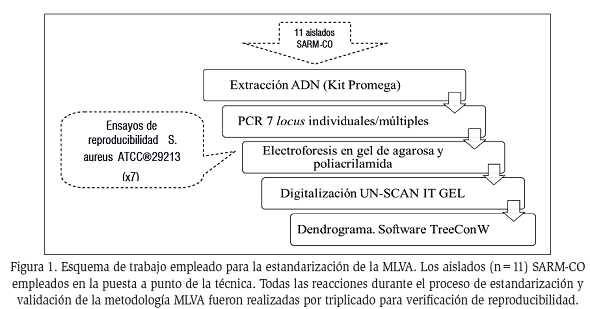
Los aislados criopreservados fueron repicados en agar sangre de carnero al 5% (BBL TM Columbia Agar Base - Becton, Dickinson and Company – BD, USA-France) e incubados a 35°C por 24-48 horas en atmósfera de 5% de CO2, para la posterior extracción del ADN. La extracción se ejecutó por dos métodos: el de ebullición, descrito por Zhang et al16 y el otro empleando el kit comercial para purificación de ADN genómico (Wizard Genomic, Promega, EEUU), siguiendo las instrucciones del fabricante (Figura 1). El ADN extraído posteriormente fue conservado a -20°C. La cantidad de ADN se estimó por lectura de la absorbancia a 260 nm con el cuantificador de ADN (Biowave DNA, WPA, UK).
Estandarización de la MLVA
Se amplificaron por PCR de forma sucesiva cada uno de los cinco pares de oligonucleótidos y luego la versión multiplex, modificando las condiciones originales de Sabat et al6 y las descritas por Rivero-Pérez et al7, como se lista a continuación.
Concentraciones óptimas para la reacción de PCR: buffer 0,8X (Bioron, Alemania), dNTPs 0,2mM (Takara, Japón), oligonucleótidos clfA-F/R 2,2 μM (Macrogen, Corea), clfB-F/R 1,5μM (Macrogen, Corea), sdrCDE-F/R 1,2μM (Macrogen, Corea), sspA-F/R 1μM (Macrogen, Corea), spAF/R 0,5μM (Macrogen, Corea), MgCl2 (Bioron, Alemania) 1,5 mM, Taq polimerasa (Bioron, Alemania) 1U, DMSO al 5% (dimetilsulfóxido). Rango de ADN empleado como molde: 20-80ng/uL, volumen total de reacción: 20uL. Las condiciones de ciclado fueron: desnaturalización inicial a 94°C por 5 minutos, 20 ciclos de desnaturalización (94°C por 30 segundos), anillamiento (58,2°C por 45 segundos) y extensión (72°C por 1,5 minuto), extensión final a 72°C por 5 minutos. Se utilizó el termociclador Esco Healthcare (Swift, Max Pro, Singapur).
Se llevó a cabo la electroforesis en gel de agarosa al 2% en buffer TBE (Tris-Borato-EDTA) 0,5% y tinción con bromuro de etidio y en gel de poliacrilamida (gel separador 7,5%, gel concentrador 5%) con tinción de nitrato de plata. Para la electroforesis en gel de poliacrilamida se sembró 7μL de producto de PCR mezclado con 2μL de loadding buffer 6% y corrida en buffer TBE al 0,5% durante 70 minutos a 800V, 200mA, 30W y potencia constante igual a 10W. En cada corrida se incluyó marcador de peso molecular de 50pb (Embiotec, Argentina). La tinción con nitrato de plata se hizo siguiendo el protocolo descrito por Sambrook y Fritsch17. Los geles de poliacrilamida, una vez teñidos, fueron secados, digitalizados con el software UN SCAN IT GEL 6.1 y analizados (Figura 1).
Los datos fueron analizados con el software TreeConW, mediante la realización de un dendograma que agrupó a los aislados en base a su similitud. Las agrupaciones fueron numeradas para su identificación y el criterio de análisis empleado corresponde a patrones MLVA que difieren en una o más bandas son consideradas como perfiles distintos6. Se efectuó la comparación de patrones MLVA obtenidos para establecer existencia de proximidad evolutiva (Figura 1)5,6.
Para comprobar la reproducibilidad de las reacciones de PCR múltiple de la técnica MLVA se incluyó la cepa S. aureus ATCC 29213 (mecA negativo y productor de una β-lactamasa débil) en cada ronda de amplificación de tal forma a verificar la amplificación de cada una de las bandas esperadas en cada reacción (n=7).

Análisis estadístico
Los datos de los aislados obtenidos de las fichas epidemiológica, microbiológica y molecular fueron introducidos, almacenados y analizados estadísticamente en una planilla electrónica empleando los softwares Microsoft Excel versión 2013 y Epiinfo versión 3.2.
Declaración sobre aspectos éticos
Se mantuvo la confidencialidad en el manejo de los datos de los aislados, ya que los mismos fueron manejados de manera estricta con códigos para su identificación. La información obtenida se manejó exclusivamente con fines científicos de carácter epidemiológico, respetando de esta manera todas las normas éticas de acuerdo con la Declaración de Helsinki.
RESULTADOS
Se obtuvo la amplificación de cada uno de los genes en forma individual sin embargo en la amplificación múltiple de los mismos algunos genes no amplificaron. Los cambios introducidos para lograr la amplificación óptima incluyeron los siguientes factores: a) calidad y cantidad de ADN molde: 40ng/uL ADN extraído por método comercial; b) condiciones de reacción: agregado de un mejorador (DMSO al 5%) y concentraciones de reactivos descritos en el apartado materiales y métodos; c) condiciones de ciclado: disminución de 5°C de la temperatura de anillamiento de los oligonucleótidos.
Para el análisis de los productos de PCR obtenidos en la técnica MLVA se realizó una electroforesis en gel de agarosa con tinción de bromuro de etidio, como indica la publicación original, pero la resolución de los fragmentos en la corrida electroforética y la sensibilidad de la tinción no fueron suficientes como para observar todas las bandas de los fragmentos de ADN generados en la amplificación. Por lo que se separaron los fragmentos de ADN por electroforesis en gel de poliacrilamida.
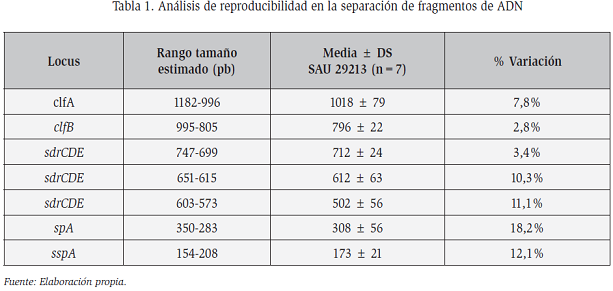
El rango probable de tamaños de productos amplificados para cada locus se estimó por análisis bioinformático empleando el software BLAST. Se alinearon los oligonucleótidos utilizados con cepas de referencia, obtenidas de la base de datos del GeneBank: NRS 100, 502A, USA300-ISMMS1 y Z172 (Tabla 1). Los productos amplificados en los once aislados y de la cepa S. aureus ATCC 29213 tenían tamaños congruentes con el rango estimado para cada locus.
Para verificar la reproducibilidad del tamaño de los productos MLVA se emplearon los datos de tamaños de productos obtenidos en las amplificaciones repetitivas (n=7) de la cepa S. aureus ATCC 29213, esta mostró reacciones completas con amplificación de las siete bandas esperadas en todas las repeticiones. Con los valores obtenidos se calculó la media y el desvío estándar del tamaño de cada producto. El valor medio de cada uno de los amplicones de la cepa S. aureus ATCC 29213 se encontró dentro del rango de tamaños esperados y se observaron variaciones de entre el 2,8 y 18,2% entre mediciones (Tabla 1).
Para la obtención del dendograma se probaron varios softwares que realizaran esta función, el TreeConW fue el elegido por ser versátil y arrojar dendogramas con datos de porcentaje de similitud entre aislados.
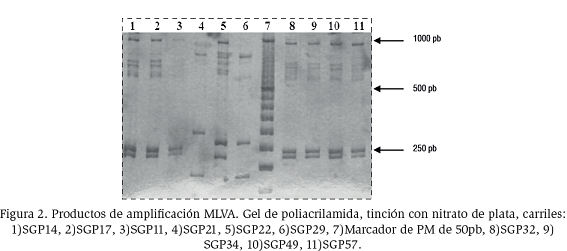
El análisis de la reproducibilidad conllevó la repetición del ensayo por triplicado, empleando siempre los mismos aislados seleccionados por poseer los datos de caracterización por técnicas moleculares como: tipificación del cassette SCCmec, spA typing y secuenciotipo por MLST. Los dendogramas obtenidos fueron exactamente iguales (100% similitud). En la figura 2 se muestra un ejemplo de electroforesis de los once aislados analizados en el estudio. La tipificación MLVA fue completa en el 100% de los aislados en estudio.
Comparación con PFGE, tipificación del cassette SCCmec, locus spa y MLST
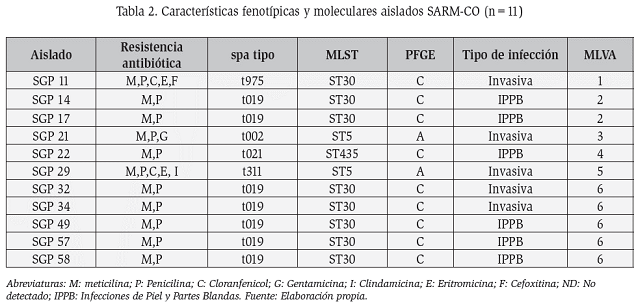
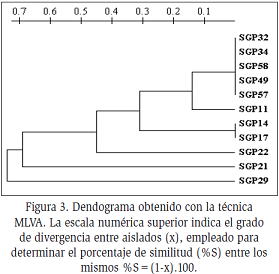
El empleo de aislados tipificados previamente para tipo de cassette SSCmec, spa typing y MLST, permitió la comparación de los resultados obtenidos por MLVA con los demás métodos. La PFGE es el gold standard para poder validarla (Tabla 2 y Figura 3). Todos los aislados presentaron cassette SCCmec tipo IV y resultaron ser portadores del gen codificante de la PVL, a excepción del SGP 21.
Se pudieron distinguir seis agrupaciones diferentes por MLVA (numeradas del uno al seis), dentro de cada agrupación todos los aislados presentaron las mismas características, tanto fenotípicas como genotípicas. La PFGE solo pudo distinguir dos perfiles (A y C) entre los once aislados estudiados. Entre aislados agrupados en un mismo perfil PFGE se observaron aún diferencias en cuanto a antibiotipo, portación de genes de virulencia, spatipo y MLST. La MLVA fue incluso capaz de distinguir estas diferencias. Aquellas agrupaciones de MLVA que abarcaron un solo aislado, presentaron resultados congruentes con otras metodologías, ya que mostraron diferentes spatipos, secuenciotipo (ST) y tipo PFGE.
DISCUSIÓN
Los productos de PCR obtenidos por cada aislado utilizando la técnica MLVA varían en tamaño y la combinación de los mismos genera un patrón de bandas característico del aislado en estudio, el cual permite la comparación con otros aislados mediante el uso de softwares informáticos. Como cualquier otro método de tipificación, la MLVA requiere un proceso de estandarización para obtener una configuración específica en el lugar que será llevado a cabo por primera vez6.
En el proceso de estandarización de la MLVA llevado a cabo en este estudio, algunas de las condiciones de reacción citadas en el artículo original de Sabat et al6 tuvieron que ser modificadas para su optimización. Se comprobó que el ADN obtenido por el método de extracción de ebullición no permitía la amplificación de la totalidad de los loci analizados, generando amplificaciones parciales. Ante esta situación se optó por aplicar un kit de extracción de ADN comercial que permitiera obtener mayor pureza del ADN para evitar interferencias en la amplificación durante la PCR múltiple, con lo que se obtuvieron óptimos resultados; incluso a diferencia de otros autores que como Rivero-Pérez et al7 indicaron que la PCR múltiple para la MLVA puede ser ejecutada exitosamente empleando ADN molde directamente de la suspensión celular. Es importante tener en cuenta que los sistemas de amplificación múltiple se caracterizan por complejas interacciones entre el molde y los diferentes oligonucleótidos presentes en la mezcla de reacción, pudiendo generar productos inespecíficos o bien inhibir la amplificación de ciertos locus. La presencia de impurezas en el ADN empleado como molde podría favorecer este último fenómeno6,8,16.
Otro factor que se ha descrito como causante de inhibición en las reacciones de PCR múltiples es la cantidad de ADN molde empleado. Tras emplear como molde un rango de concentraciones de ADN de 20ng a 80ng, se descubrió que los mejores resultados se obtenían con 40ng de ADN por reacción. Otros ajustes llevados a cabo con el afán de mejorar el rendimiento y la eficiencia de la reacción incluyeron la disminución de la concentración del buffer, la temperatura de anillamiento y el agregado de DMSO en la mezcla de reacción; todos estos contribuyeron de algún modo para obtener las condiciones óptimas de reacción.
Con los datos obtenidos por electroforesis se realizó un dendograma que agrupó a los aislados con base en su similitud mediante el uso del software TreeConW, que presenta la ventaja frente a otros de arrojar datos de porcentaje de similitud entre aislados.
La tipificación MLVA se logró en el 100% de los aislados en estudio, un nivel excelente comparado con otros estudios que reportan niveles de tipificación de entre el 76% al 100%, por no lograr la amplificación de la totalidad de los locus originando perfiles parciales, probablemente por mutaciones en las regiones de anillamiento de los oligonucleótidos4-6,18.
Mediante la amplificación repetida de la cepa de referencia S. aureus ATCC 29213, se observó la amplificación de la totalidad de los locus en los siete experimentos ejecutados. Así mismo el tamaño de los productos obtenidos para cada locus generó valores con mínimas variaciones de tamaño. Las desviaciones más acentuadas se observaron en aquellas bandas de menor tamaño y que presentaron mayor migración en el gel de poliacrilamida. Estas variaciones son propias de la técnica de electroforesis y no afectaron la posición de los aislados dentro de cada dendograma, porque se observó la misma variación en todos los amplicones dentro de un mismo gel, respecto al marcador molecular incluido en el gel. Esto denota un alto nivel de reproducibilidad de la reacción MLVA, coincidiendo con lo descrito por otros autores5,7,19.
El empleo de cepas previamente tipificadas por MLST, PFGE, locus spa y cassette SCCmec, permitió validar de forma comparativa el agrupamiento generado por la MLVA. El número de muestras (n=11) del estudio fue suficiente para validar la técnica, teniendo en cuenta que todas ellas fueron sometidas a todos los métodos de tipificación empleados para validar la MLVA, por lo que la N muestral no es una limitación para la extrapolación de los resultados hacia otros aislados no incluidos en este estudio. Aun así, sería interesante en un futuro próximo ensayar la técnica con SARM aislados del hospital.
Los aislados que fueron agrupados como idénticos por MLVA presentaron resultados congruentes con la totalidad de otras técnicas moleculares, lo que demuestra que la MLVA tiene la capacidad de identificar aislados idénticos y agruparlos de forma comparable con MLST, spa typing y tipo de cassette SCCmec. Algunos estudios, como el publicado por Schouls et al4, que comparan estas metodologías con un número mayor de aislados en estudio (n=1681), indican incluso que la MLVA tiene mayor poder discriminatorio que la tipificación del locus spA y MLST y posee mayor rendimiento y es más fácil de llevarse a cabo que este último.
Es importante reconocer que debido a que la metodología MLVA analiza de forma puntual la variación en siete regiones genéticas, puede darse la situación en que estas sean idénticas, pero la diferencia entre aislados se encuentre en otras regiones del genoma. Por tanto MLVA agruparía a estos aislados como idénticos cuando solo son similares pero diferentes, y para demostrar esta diferencia se requiere el uso de PFGE. Aun así, la capacidad discriminatoria de la metodología MLVA no es despreciable e incluso es congruente con el uso combinado de MLST, spa typing y tipo de cassette SCCmec. En este estudio, la MLVA demostró ser aún más sensible que la PFGE para distinguir entre aislados que presentaron patrones de PFGE idénticos, situación que ya había sido descrita por Moser et al20, Tenover et al19 y comprobada por Holmes et al18 en cuyo estudio fue incluso capaz de distinguir entre aislados con un mismo PFGE por presentar variaciones en la sensibilidad a un antibiótico.
Claramente la metodología MLVA cumple todos los criterios para ser utilizada como método de tipificación de S. aureus, y es adaptable a estudios epidemiológicos a gran escala de infecciones estafilocócicas. Algunos autores remarcan la notable sensibilidad del método, indicando que puede ser empleado para tipificación de S. aureus directamente a partir de muestras clínicas, sin necesidad de partir del cultivo, en estudios que requieran una respuesta rápida como análisis de brotes6. Podría emplearse como método de screening rápido y fácil de realizar, en el cual solo deban someterse a PFGE (análisis del genoma completo) aquellos aislados que resulten agrupados como idénticos por MLVA para descartar brotes; con este tipo de estrategias no sólo se obtendrían resultados en menor tiempo y a menor costo, sino que se podría optimizar el uso de recursos en el empleo de PFGE que hasta ahora es muy restringido en Paraguay.
Este estudio demuestra la habilidad de la MLVA para identificar aislados genéticamente diferentes y agrupar los relacionados estrechamente entre sí, que puedan tratarse del mismo clon. Sin embargo, la metodología MLVA, al igual que otras metodologías validadas como la MLST y la tipificación del locus spA, puede agrupar como aislados idénticos a aquellos estrechamente relacionados, por lo que precisaría en algunos casos ser verificada por la PFGE, que analiza el genoma completo, lo que aumenta su poder discriminatorio4,7,21,22.
Los datos obtenidos a través de la metodología MLVA indican que esta cumple con todos los criterios de un método de tipificación útil para aislados SARM-CO. Frente a las metodologías validadas mencionadas, MLVA presenta las ventajas de tener bajo costo, ser simple y requerir poco tiempo para su ejecución, lo que hace que esté disponible en el país (Paraguay) para ser llevada a cabo de forma rutinaria, principalmente como herramienta epidemiológica molecular para monitoreo de brotes locales6,7,18.
DECLARACIÓN SOBRE CONFLICTO DE INTERESES
El presente estudio fue realizado con financiación parcial del Consejo Nacional de Ciencia y Tecnología del Paraguay (CONACYT) y mediante fondos propios del grupo de investigación.
Los autores declaran ser independientes respecto a las instituciones financiadoras y de apoyo, y durante la ejecución del trabajo o la redacción del manuscrito no han incidido intereses o valores distintos a los que usualmente tiene la investigación.
REFERENCIAS BIBLIOGRÁFICAS
1. Collery MM, Smyth DS, Twohig JM, Shore AC, Coleman DC, Smyth CJ. Molecular Typing of Nasal Carriage Isolates of Staphylococcus aureus from an Irish University Student Population Based on Toxin Gene PCR, Agr Locus Types and Multiple Locus, Variable Number Tandem Repeat Analysis. J Med Microbiol. 2008; 57(3):348-58.
2. Paganini M, Della LP, Muller OB, Ezcurra G, Uranga M, Aguirre C, et al. Infecciones por Staphylococcus aureus resistente a meticilina adquiridas en la comunidad en niños antes sanos y en niños relacionados al hospital en la Argentina. Rev Chil Infectol. 2009; 26(5):406-12.
3. Pertuz-Meza Y, Perez-Quintero C, Pabón-Varela Y. Aspectos epidemiológicos de la sepsis, en unidades de cuidados intensivos Santa Marta, Colombia. Duazary. 2016; 13(2):126-32.
4. Schouls LM, Spalburg EC, van Luit M, Huijsdens XW, Pluister GN, van Santen-Verheuvel MG, et al. Multiple-Locus Variable Number Tandem Repeat Analysis of Staphylococcus aureus: Comparison with Pulsed-Field Gel Electrophoresis and spa-Typing. PLoS ONE. 2009; 4(4).
5. Malachowa N, Sabat A, Gniadkowski M, KrzysztonRussjan J, Empel J, Miedzobrodzki J, et al. Comparison of Multiple-Locus Variable-Number Tandem-Repeat Analysis with Pulsed-Field Gel Electrophoresis, spa Typing, and Multilocus Sequence Typing for Clonal Characterization of Staphylococcus aureus Isolates. J Clin Microbiol. 2005; 43(7):3095-100.
6. Sabat A, Krzyszton-Russjan J, Strzalka W, Filipek R, Kosowska K, Hryniewicz W, et al. New Method for Typing Staphylococcus aureus Strains: MultipleLocus Variable-Number Tandem Repeat Analysis of Polymorphism and Genetic Relationships of Clinical Isolates. J Clin Microbiol. 2003; 41(4):1801-4.
7. Rivero-Pérez B, Pérez-Roth E, Méndez-Álvarez S. Evaluation of Multiple-Locus Variable-Number TandemRepeat Analysis for Typing a Polyclonal HospitalAcquired Methicillin-Resistant Staphylococcus aureus Population in an Area Where Such Infections Are Endemic. J Clin Microbiol. 2010; 48(8):2991-4.
8. Pérez-Roth E, Claverie-Martín F, Villar J, MéndezÁlvarez S. Multiplex PCR for Simultaneous Identification of Staphylococcus aureus and Detection of Methicillin and Mupirocin Resistance. J Clin Microbiol. 2001; 39(11):4037-41.
9. Mayor L, Ortellado J, Menacho C, Lird G, Courtier C, Gardon C, et al. Molecular Characterization of Methicillin-Resistant Staphylococcus aureus Isolates Collected in Asunción, Paraguay. J Clin Microbiol. 2007; 45(7):2298-300.
10. Weiler N, Leotta GA, Zarate MN, Manfredi E, Alvarez ME, Rivas M. Brote de intoxicación alimentaria asociado al consumo de leche ultrapasteurizada en la República del Paraguay. Revista Argentina de Microbiología. 2011; 43:33-6.
11. Rodríguez F, Carpinelli L, Basualdo W, Castro H, Quiñónez B, Arguello R, et al. Frecuencia de genes que codifican factores de virulencia en Staphylococcus aureus aislados de niños que concurrieron al Hospital General Pediátrico Niños de Acosta Ñú, durante el año 2010. Mem IICS. 2015; 13(1):58-66.
12. Shopsin B, Gomez M, Montgomery SO, Smith DH, Waddington M, Dodge DE, et al. Evaluation of Protein A Gene Polymorphic Region DNA Sequencing for Typing of Staphylococcus aureus Strains. J Clin Microbiol. 1999; 37(11):3556-63.
13. Kondo Y, Ito T, Ma XX, Watanabe S, Kreiswirth BN, Etienne J, et al. Combination of Multiplex PCRs for Staphylococcal Cassette Chromosome mec Type Assignment: Rapid Identification System for mec, ccr, and Major Differences in Junkyard Regions. Antimicrob Agents Chemother. 2007; 51(1):264-74.
14. Enright MC, Day NPJ, Davies CE, Peacock SJ, Spratt BG. Multilocus Sequence Typing for Characterization of Methicillin-Resistant and Methicillin-Susceptible Clones of Staphylococcus aureus. J Clin Microbiol. 2000; 38(3):1008-15.
15. Chung M, de Lencastre H, Matthews P, Tomasz A, Adamsson I, Aires de Sousa M, et al. Molecular typing of methicillin-resistant Staphylococcus aureus by pulsed-field gel electrophoresis: comparison of results obtained in a multilaboratory effort using identical protocols and MRSA strains. Microb Drug Resist Larchmt N. 2000; 6(3):189-98.
16. Zhang K, Sparling J, Chow BL, Elsayed S, Hussain Z, Church DL, et al. New quadriplex PCR assay for detection of methicillin and mupirocin resistance and simultaneous discrimination of Staphylococcus aureus from coagulase-negative staphylococci. J Clin Microbiol. 2004; 42(11):4947-55.
17. Sambrook J, Fritsch EF. Molecular Cloning: A Laboratory Manual. Nueva York: Cold Spring Harbor Laboratory Press; 1989.
18. Holmes A, Edwards GF, Girvan EK, Hannant W, Danial J, Fitzgerald JR, et al. Comparison of Two Multilocus Variable-Number Tandem-Repeat Methods and PulsedField Gel Electrophoresis for Differentiating Highly Clonal Methicillin-Resistant Staphylococcus aureus Isolates. J Clin Microbiol. 2010; 48(10):3600-7.
19. Tenover FC, Vaughn RR, McDougal LK, Fosheim GE, McGowan JE. Multiple-Locus Variable-Number Tandem-Repeat Assay Analysis of Methicillin-Resistant Staphylococcus aureus Strains. J Clin Microbiol. 2007; 45(7):2215-9.
20. Moser SA, Box MJ, Patel M, Amaya M, Schelonka R, Waites KB. Multiple-locus variable-number tandemrepeat analysis of meticillin-resistant Staphylococcus aureus discriminates within USA pulsed-field gel electrophoresis types. J Hosp Infect. 2009; 71(4):333-9.
21. Bustamante AV, Lucchesi PMA, Parma AE. Molecular characterization of Verocytotoxigenic Escherichia coli O157:H7 isolates from Argentina by Multiple-Loci VNTR Analysis (MLVA). Braz J Microbiol. 2009; 40(4):927-32.
22. Blomfeldt A, Hasan AA, Aamot HV. Can MLVA Differentiate among Endemic-Like MRSA Isolates with Identical Spa-Type in a Low-Prevalence Region? PloS One. 2016;11(2):e0148772.
Para citar este artículo: Rodríguez F, Fernández S, Haim S, Mollerach M, Basualdo W, Castro H, et ál. Estandarización del análisis multi-locus de número variable de repeticiones en tándem para el estudio de Staphylococcus aureus resistentes a meticilina aislados de la comunidad en Paraguay. Duazary. 2017 julio; 14 (2): 131 - 140. Doi: http://dx.doi. org/10.21676/2389783X.1971
Recibido en octubre 06 de 2016
Aceptado en enero 23 de 2017
Publicado en línea en mayo 15 de 2017